使用Multiwfn結合CP2K通過NCI和IGM方法圖形化考察固體和表面的弱相互作用
使用Multiwfn結合CP2K通過NCI和IGM方法圖形化考察固體和表面的弱相互作用
文/Sobereva@北京科音
First release: 2021-Feb-27 Last update: 2022-Mar-8
1 前言
2010年提出的Noncovalent interaction (NCI)分析,也即Reduced density gradient (RDG)分析,是如今使用極為普遍的圖形化考察化學體系中弱相互作用的方法,筆者寫過諸多相關文章介紹原理和實現,還錄了操作演示視頻,如下所示,故本文對基本原理不再累述,并假定讀者已經具備了背景知識
? 《使用Multiwfn圖形化研究弱相互作用》(http://www.shanxitv.org/68)
? 《用Multiwfn+VMD做RDG分析時的一些要點和常見問題》(http://www.shanxitv.org/291)
? 《通過鍵級曲線和ELF/LOL/RDG等值面動畫研究化學反應過程》(http://www.shanxitv.org/200)
? 《使用Multiwfn做NCI分析展現分子內和分子間弱相互作用》(演示視頻。https://www.bilibili.com/video/av71561024)
? Multiwfn手冊3.23.1節
? 《使用Multiwfn研究分子動力學中的弱相互作用》(http://www.shanxitv.org/186)
筆者開發的Multiwfn是做NCI分析最流行、最好用的程序。但以前的版本主要面向分子體系,基于Gaussian、ORCA等量子化學程序產生的波函數進行分析。對于周期性體系往往也可以通過團簇模型結合Multiwfn做NCI分析,比如《使用量子化學程序基于簇模型計算金屬表面吸附問題》(http://www.shanxitv.org/540)里對銀團簇表面吸附苯做了NCI分析、在Multiwfn原文J. Comput. Chem., 33, 580 (2012)里截取了尿素團簇做了NCI分析,但是還是有很多情況不便于或者幾乎不適合用簇模型描述。為了讓周期性體系的NCI分析可以非常容易地實現、使這個方法能考察更廣泛的體系,從2021年2月更新的Multiwfn開始,Multiwfn也支持了結合CP2K程序產生的波函數做NCI分析,這使得圖形化考察固體內部和表面的弱相互作用極其容易,將在本文進行詳細示例。看了第3節的例子后你會發現計算過程真是巨簡單。本文也會提及很多讀者應了解的注意事項,以便在實際研究中遇到問題時恰當變通。
另外,Independent gradient model (IGM)也是非常重要的圖形化研究弱相互作用的方法,相對于NCI的關鍵好處是可以很靈活地自定義片段,能只顯示片段間的弱相互作用而不會受到片段內的干擾。此外,Multiwfn做IGM分析時還可以給出諸多定量指標便于討論。Multiwfn做IGM分析在此文有詳述:《通過獨立梯度模型(IGM)考察分子間弱相互作用》(http://www.shanxitv.org/407)。如今Multiwfn也將IGM分析擴展到了周期性體系,將在下文示例。本文還會順帶提及筆者提出的IGM方法的改良,即IGM based on Hirshfeld partition (IGMH),比IGM圖像效果明顯好得多,詳見《使用Multiwfn做IGMH分析非常清晰直觀地展現化學體系中的相互作用》(http://www.shanxitv.org/621)。
值得一提的是,Multiwfn還支持筆者提出的IRI方法,可以同時把弱相互作用和化學鍵作用區域直觀地展現,很有實用價值,在《使用IRI方法圖形化考察化學體系中的化學鍵和弱相互作用》(http://www.shanxitv.org/598)中有專門的介紹,并且給了計算孤立體系和計算周期性體系的例子,強烈建議一看!
對一般情況,筆者強烈建議用后來提出的IRI代替NCI,能明顯展現更多有價值的信息;也強烈建議用后來提出的IGMH代替IGM,圖像效果好得多,且物理意義更嚴格。IRI和NCI的分析操作幾乎完全相同,而IGMH和IGM的分析操作幾乎完全相同。 在《一篇最全面介紹各種弱相互作用可視化分析方法的文章已發表!》(http://www.shanxitv.org/667)中介紹的筆者的綜述文章中對上述可視化分析方法有非常全面深入的介紹,里面也展示了用于周期性體系的例子,推薦閱讀和引用。
Multiwfn可以在官網http://www.shanxitv.org/multiwfn免費下載,不熟悉Multiwfn的話注意參看《Multiwfn FAQ》(http://www.shanxitv.org/452)和《Multiwfn入門tips》(http://www.shanxitv.org/167)。請讀者務必使用2021-Feb-25日及之后更新的Multiwfn以確保情況和下文內容相符。本文例子中周期性體系的DFT計算部分使用CP2K 8.1版實現,安裝方法見《CP2K第一性原理程序在CentOS中的簡易安裝方法》(http://www.shanxitv.org/586)。CP2K結合Multiwfn可以非常容易地使用,見《使用Multiwfn非常便利地創建CP2K程序的輸入文件》(http://www.shanxitv.org/587)。本文繪圖使用VMD 1.9.3版,可以在http://www.ks.uiuc.edu/Research/vmd/免費下載。
下文涉及的所有文件都可以在http://www.shanxitv.org/attach/588/file.rar下載。cub文件比較大所以就沒有提供了。
2 輸入文件
在給出具體例子之前先介紹一下Multiwfn做NCI、IGM分析都需要用什么輸入文件。
后記:關于這部分知識,更多、更詳細的介紹見《詳談使用CP2K產生給Multiwfn用的molden格式的波函數文件》(http://www.shanxitv.org/651)。
2.1 普通NCI分析用的輸入文件
普通形式的NCI分析需要基于電子波函數計算電子密度及其導數。CP2K產生的molden文件記錄了Gaussian型基函數描述的電子波函數,Multiwfn可以基于它做各種波函數分析,也包括NCI分析。CP2K產生molden文件的方法在《使用Multiwfn非常便利地創建CP2K程序的輸入文件》(http://www.shanxitv.org/587)的3.1節已經示例過。如果你習慣自己寫CP2K輸入文件的話,是在&DFT字段里加上以下內容
&PRINT
&MO_MOLDEN
NDIGITS 8
GTO_KIND SPHERICAL
&END MO_MOLDEN
&END PRINT
這樣CP2K計算完畢后就會在當前目錄下產生一個.molden文件。
CP2K產生的molden文件還不能直接用于做周期性體系的NCI分析,因為標準的molden格式里沒有晶胞信息。為了能讓molden文件給Multiwfn提供晶胞信息,我給molden格式做了擴展,定義了[Cell]字段。用文本編輯器打開molden文件后,需手動在第二行插入[Cell]并在下面三行定義晶胞的三個平移矢量(埃),下面是個例子
[Molden Format]
[Cell]
7.13358000 0.00000000 0.00000000
0.00000000 7.13358000 0.00000000
0.00000000 0.00000000 7.13358000
[Atoms] AU
C 1 4 0.000000 0.000000 0.000000
C 2 4 1.685064 1.685064 1.685064
C 3 4 0.000000 3.370128 3.370128
...略
讓Multiwfn載入這樣修改過的molden文件后,晶胞信息就會被讀入,文件載入完畢后屏幕上就可以看到晶胞信息的提示了:
Cell information:
Translation vector 1 X= 13.480512 Y= 0.000000 Z= 0.000000 Bohr
Translation vector 2 X= 0.000000 Y= 13.480512 Z= 0.000000 Bohr
Translation vector 3 X= 0.000000 Y= 0.000000 Z= 13.480512 Bohr
Cell volume: 2449.7350 Bohr^3 ( 363.0134 Angstrom^3 )
有一點需要注意的是,如今CP2K計算普遍使用MOLOPT基組,這是個完全廣義收縮的基組,而Multiwfn的計算代碼并未對廣義收縮的基組進行專門的優化,因此使用MOLOPT基組得到的波函數在Multiwfn里計算耗時會比較高。如果你有個像樣的服務器那無所謂,但如果只有個個人PC,算NCI的格點數據的時候對稍大的體系會比較吃力。我一般建議用比如6-31G*、6-311G**之類基本像樣的而且是片段收縮的全電子基組在CP2K里做個GAPW單點計算來得到NCI分析用的molden文件,比起用DZVP-MOLOPT-SR-GTH基組做GPW計算得到的molden文件在NCI分析耗時上能降低好幾倍。另外,用全電子基組比起用MOLOPT這樣的贗勢基組還有個好處是內核電子被明確表達了。NCI分析對基組不敏感,一般用6-31G*就足夠了,用更大基組也不會發現結果有什么可查覺的改變。
如果由于特殊情況,你就是想用諸如MOLOPT贗勢基組在Multiwfn里做NCI等波函數分析,那么應當再手動改一下molden文件,使得Multiwfn明確知道各個原子的價電子數是多少(指的是使用此贗勢時原子在孤立存在時的價電子數。和原子在當前體系里的實際價電子數沒任何關系),這有兩個必要性:
? molden文件里并沒有明確記錄原子的價電子數,這導致Multiwfn算原子電荷的時候不知道實際上當前原子的核電荷數是多少,因此給出的原子電荷是不合理的。
? 讓Multiwfn知道被贗勢贗化的電子數的話,Multiwfn會通過自帶的EDF內核電子密度數據庫提供內核電子密度,此時做純粹基于電子密度的各種分析時結果會較接近于全電子基組的情況,即更為真實。關于這點詳見《在贗勢下做波函數分析的一些說明》(http://www.shanxitv.org/156)。
讓Multiwfn知道各個原子價電子數是多少,可以通過以下兩種修改molden文件的做法實現
(1)把元素名后頭的元素序號改成價電子數。比如molden文件的[Atoms]字段某一行原本是
C 3 6 0.000000 0.000000 0.000000
如果贗勢贗化了碳的1s的兩個電子,即價電子數為4,則改為
C 3 4 0.000000 0.000000 0.000000
(2)更方便的做法是直接在[Atoms]字段之前加入[Nval]字段,這是我對molden格式的擴展。例如插入以下內容
[Nval]
C 4
O 6
就告訴Multiwfn所有的碳都是4個價電子,氧是6個價電子。加入了[Nval]和[Cell]字段的一個例子見Multiwfn自帶的examples\PBC\CP2K_diamond_2x2_DZVP-MOLOPT.inp。
注意CP2K在考慮k點的時候不能產生molden文件,Multiwfn本身也不支持考慮k點,所以對于小晶胞體系必須用足夠大的超胞來計算。
實際上Multiwfn還可以基于Gaussian程序的PBC計算產生的fch文件做周期性體系的波函數分析。但對于二維、三維周期性體系,Gaussian的這個功能幾乎什么都算不動,毫無實用性,這里就不提了。Multiwfn也可以基于含有晶胞平移矢量信息的mwfn格式(https://doi.org/10.26434/chemrxiv.11872524)做周期性體系的波函數分析,但CP2K目前版本產生不了。
2.2 準分子近似的NCI分析以及IGM分析用的輸入文件
還有一種NCI分析是基于準分子近似(promolecular approximation)實現的,即使用原子自由狀態電子密度簡單疊加產生的近似的分子電子密度(準分子密度,promolecular density)進行計算。對于周期性體系的準分子近似的NCI分析,只需要給Multiwfn提供含有原子坐標和晶胞信息的文件作為輸入文件即可,比如cif文件、Gaussian的PBC任務的gjf文件、含有CRYST1字段的pdb文件等,詳見《使用Multiwfn非常便利地創建CP2K程序的輸入文件》(http://www.shanxitv.org/587)的第2節。
準分子近似的NCI計算耗時只是普通NCI分析的一個零頭。而且由于只需要結構信息,用任何第一性原理程序優化坐標/晶胞都可以,也可以直接用實驗的cif文件。但其結果合理性比普通的NCI略差,盡管通常來說定性一致。一般來說能算得動普通的NCI分析的時候就不建議用準分子近似。
IGM分析需要的輸入文件和準分子近似的NCI分析完全一樣,因為IGM分析也是完全依賴于幾何結構和Multiwfn內置的自由狀態原子密度實現的。
3 周期性體系的NCI分析示例
3.1 金剛石中的位阻作用
3.1.1 普通NCI分析
此例用NCI分析考察金剛石中的位阻作用。首先基于晶體結構,用Multiwfn產生一個做PBE/6-31G*單點計算并輸出molden文件的CP2K輸入文件,使用2*2*2的超胞。為此,用Multiwfn載入本文文件包里的金剛石的conventional cell的文件diamond.cif,然后輸入
cp2k //創建CP2K輸入文件
diamond_222.inp //要產生的CP2K輸入文件名
2 //選擇基組
10 //6-31G*
-2 //要求產生molden文件
-11 //幾何操作
19 //構建超胞
2 //第1個方向重復的次數
2 //第2個方向重復的次數
2 //第3個方向重復的次數
-10 //返回
0 //產生輸入文件
現在diamond_222.inp文件被產生在了當前目錄下,CP2K算完后就有了diamond_222-MOS-1_0.molden。用文本編輯器打開它,然后把diamond_222.inp里的晶胞信息復制進去,此時此文件開頭部分內容為
[Molden Format]
[Cell]
7.13358000 0.00000000 0.00000000
0.00000000 7.13358000 0.00000000
0.00000000 0.00000000 7.13358000
[Atoms] AU
C 1 6 0.000000 0.000000 0.000000
C 2 6 0.000000 3.370128 3.370128
...略
啟動Multiwfn,載入diamond_222-MOS-1_0.molden,然后輸入
20 //圖形化分析弱相互作用
1 //NCI分析
9 //借助晶胞信息定義格點
[按回車] //使用0,0,0作為盒子原點。這里說的盒子是指被計算的格點均勻分布的區域,后同
[按回車] //使用晶胞的三個平移矢量的長度作為盒子三個邊的長度
0.2 //格點間距
現在Multiwfn就開始計算NCI分析用的RDG和sign(λ2)rho這兩個函數的格點數據了。在筆者的2*E5-2696v3 36核服務器上僅僅11秒就算完了。之后選3把這兩個函數的格點數據分別導出為當前目錄下的func2.cub和func1.cub。將這兩個cub文件以及Multiwfn自帶的examples目錄下的RDGfill.vmd都拷到VMD目錄下,然后啟動VMD,在VMD的文本窗口輸入source RDGfill2.vmd執行此繪圖腳本,再輸入pbc box來繪制出盒子。之后在Graphics - Representation里選擇正處于CPK風格的那個representation,通過界面上的Sphere Scale把原子球尺寸調成0.7。此時的sign(λ2)rho著色的RDG等值面圖,也即一般說的NCI圖,如下所示
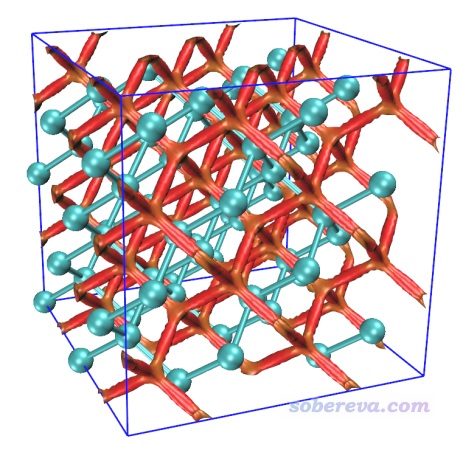
可見圖像效果非常理想。紅色的等值面在金剛石中呈網狀分布,體現出金剛石里面有顯著的位阻作用。讀者有興趣的話可以通過相同的方法去對硅也繪制這種圖,你會發現硅當中也有網狀的等值面,但顏色是綠色的,體現出由于硅的孔洞相對較大,孔洞中的位阻互斥就沒那么強了。
通常做周期性體系NCI分析時格點都按照此例方式來設即可,這樣格點會均勻分布在整個盒子里,從而把體系所有地方的弱相互作用都圖形化展現出來。格點間距應根據需要恰當設置,想要更光滑的等值面就把格點間距設得更小。注意在盒子范圍固定的情況下,計算耗時、cub文件的尺寸都和格點間距的三次方呈反比。此例如果把格點間距設成更小的0.13,你會發現得到的等值面上完全看不出任何鋸齒,但耗時會增加為原先的(0.2/0.13)^3=3.6倍。
NCI分析中經常涉及到繪制RDG與sign(λ2)rho之間的散點圖,做法在第1節里提到的筆者的RDG博文中,以及《繪制有填色效果的用于弱相互作用分析的RDG散點圖的方法》(http://www.shanxitv.org/399)中都做了介紹。周期性體系的NCI分析也可以這么繪制散點圖,這里就不再演示了,這和分析孤立體系的操作沒有任何區別。
3.1.2 準分子近似的NCI分析
現在再演示一下如何對金剛石超胞做準分子近似的NCI分析。用上一節的diamond_222.inp或者diamond_222-MOS-1_0.molden當做Multiwfn的輸入文件都可以,因為它們都能給Multiwfn提供這種分析所需要的原子坐標和晶胞信息。這里用原胞diamond.cif作為輸入文件進行演示,請大家舉一反三。
啟動Multiwfn,載入diamond.cif,然后輸入
300 //其它功能(Part 3)
7 //對體系進行幾何操作
19 //構建超胞
2
2
2
-10 //返回
0 //返回主菜單。現在內存里記錄的金剛石的原胞已經成了2*2*2的超胞了
20 //圖形化分析弱相互作用
2 //準分子近似的NCI分析
9 //設置格點。同前,不再解釋
[按回車]
[按回車]
0.2 //格點間距(Bohr)
你會充分體會到準分子近似的NCI分析極快,一眨眼的功夫格點數據就算完了。之后選3導出cub文件,將導出的func1.cub、func2.cub以及Multiwfn自帶的examples目錄下的RDGfill_pro.vmd都復制到VMD目錄下。啟動VMD,在文本窗口里輸入source RDGfill_pro.vmd,然后輸入pbc box顯示盒子。之后在Graphics - Representation界面里改一下CPK原子球尺寸,然后選Isosurface那個representation,把Isovalue改成0.2并按回車。此時圖像如下,可見和普通NCI分析得到的圖像特征定性一致。
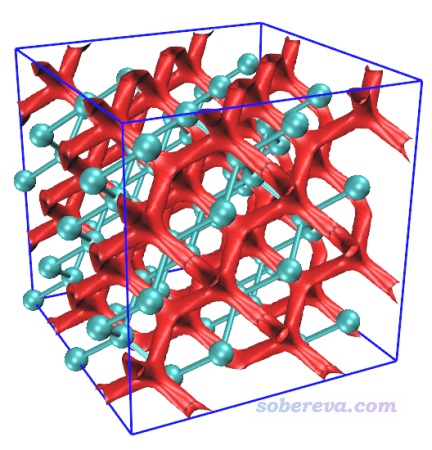
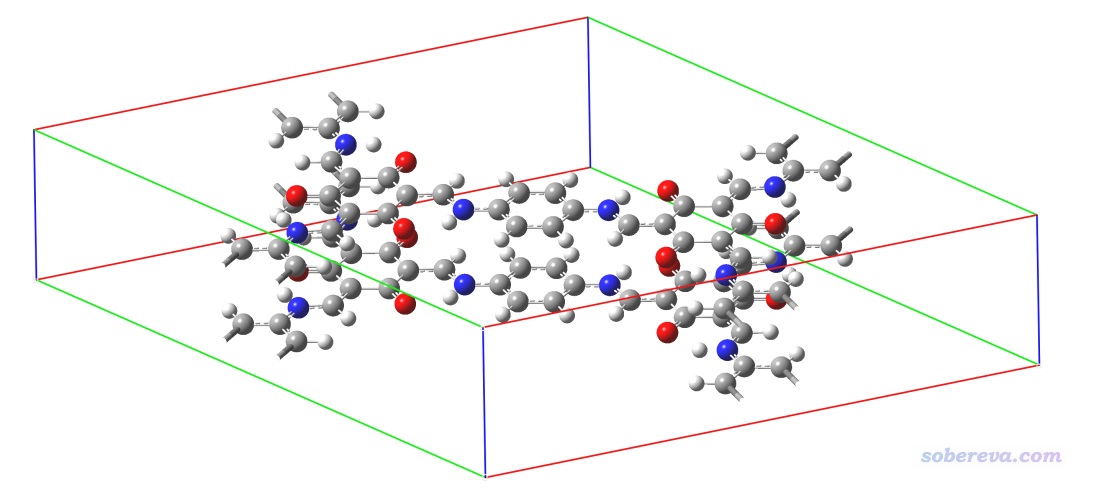
3.2 共價有機框架化合物(COF)中的弱相互作用
此例將對下面這個COF晶胞中的弱相互作用通過NCI方法進行圖形化展現。
此體系的cif文件是本文文件包里的COF_12000N2.cif。這個晶胞已經很大了,顯然沒必要再弄成超胞。如《實驗測定分子結構的方法以及將實驗結構用于量子化學計算需要注意的問題》(http://www.shanxitv.org/569)一文所述,X光衍射得到的晶體結構中氫的位置通常是不可靠的,所以本例我們在做NCI分析前用CP2K先優化一下氫原子的位置。
啟動Multiwfn并載入COF_12000N2.cif,然后輸入
COF_12000N2.cif
cp2k
COF_opt.inp
-2 //要求產生molden文件
-1 //設置任務類型
3 //優化結構但不優化晶胞
9 //設置原子凍結
optH //只優化氫原子
2 //選擇基組
10 //6-31G*
0 //產生輸入文件
這樣就得到了PBE/6-31G*只優化COF中氫原子位置的CP2K輸入文件COF_opt.inp。運行之,在36核服務器上十來分鐘就算完了。之后可以看到一批以COF_opt-MOS-1_為開頭的molden文件,它們記錄了優化過程中各幀的波函數。其中序號最大的是COF_opt-MOS-1_4.molden,這是最后優化完的結構下的波函數文件。將COF_opt.inp里的晶胞信息手動加入到COF_opt-MOS-1_4.molden文件里作為[Cell]字段。
用Multiwfn載入COF_opt-MOS-1_4.molden,然后按照與3.1節完全相同的操作步驟讓Multiwfn做普通的NCI分析。使用0.2 Bohr格點間距時,在筆者的36核服務器上花了不到一分鐘算完。然后用前例說的RDGfill2.vmd腳本在VMD里對Multiwfn導出的func1.cub和func2.cub作圖,結果如下
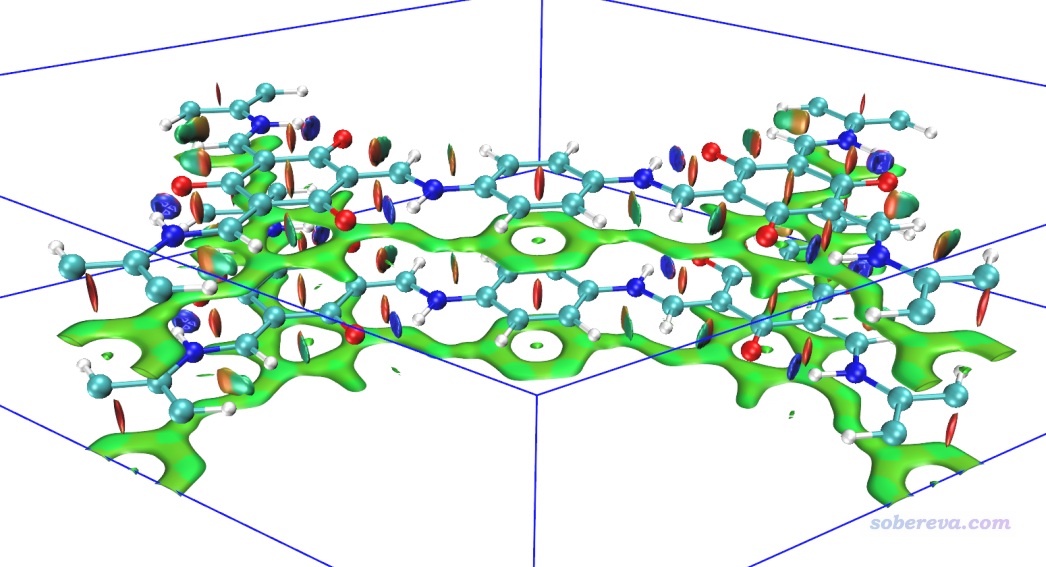
上圖非常清晰直觀地將COF晶胞中的兩層結構間的大面積pi-pi堆積展現了出來。苯環中央的紅色錐形等值面展現了環中的位阻作用。當前體系中強烈的N-H...O內氫鍵被非常藍的圓片型等值面表現了出來。
上圖一些等值面還不算特平滑,有的等值面邊緣鋸齒有點明顯,將格點間距降到0.15會明顯更好看,但耗時會多一倍多,每個cub文件增大到90兆。另外,上圖左側和右側的N-H...O內氫鍵的圓片型等值面中間凹陷了一些,像缺了一塊,這是因為這個內氫鍵很強,在作用區域有的地方電子密度超過了0.05 a.u.,而Multiwfn默認會把電子密度超過0.05 a.u.區域的RDG設為100來避免顯示出強作用區域(如化學鍵)的等值面。為解決這個問題,可以把Multiwfn目錄下的settings.ini中的RDG_maxrho從默認0.05設為改大到0.06 a.u.,這樣重新計算和繪制后就不會看到這個強內氫鍵的RDG等值面中間凹陷(被屏蔽掉)一塊了。
如果你想把上圖中下方的相鄰晶胞(-Z晶胞)的結構和RDG等值面也顯示出來,就進入Graphics - Representation界面,對每個Representation都進入Periodic標簽頁并勾選上-Z,即可看到下圖
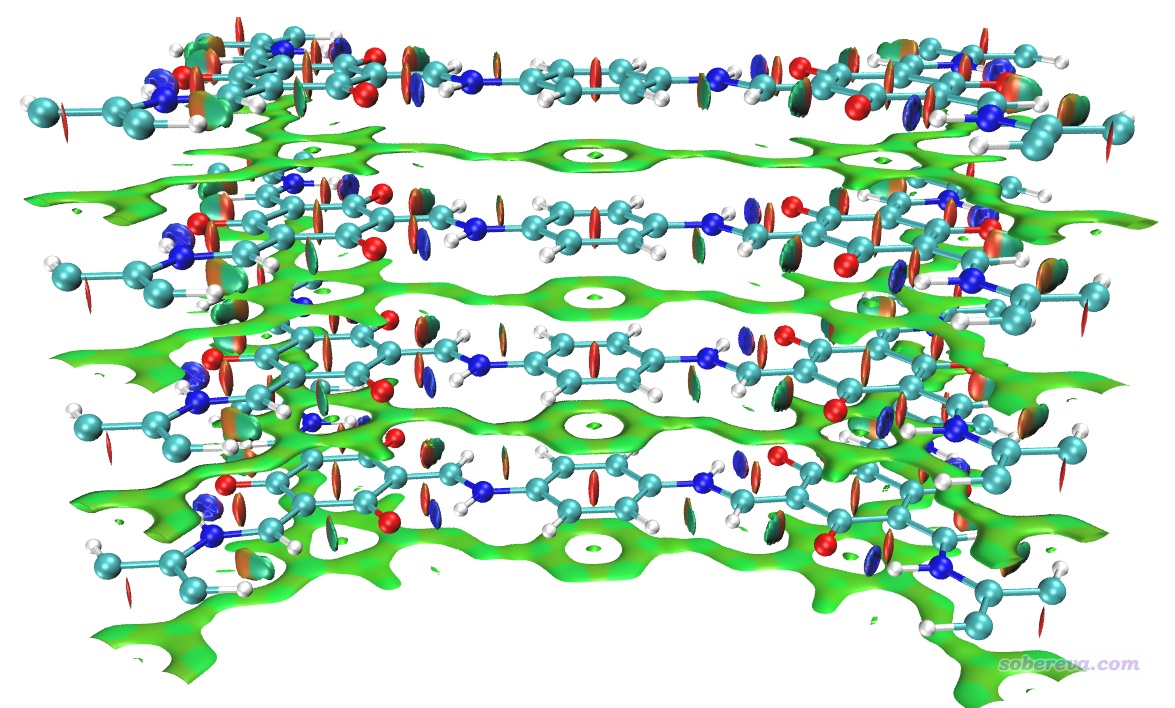
在之前不顯示相鄰鏡像的NCI圖中,我們看到在當前晶胞的底端也出現了大面積綠色等值面,這是這部分結構與-Z鏡像晶胞的上層部分的相互作用所導致的,看起來很礙眼,怎么避免顯示出來呢?在Multiwfn中有兩種做法可以實現:
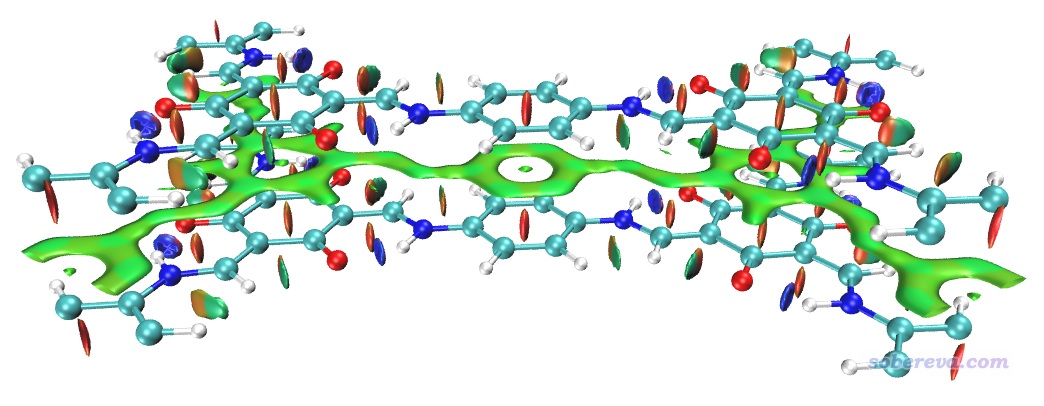
(1)讓盒子起點位置的Z坐標稍微大于0,并讓盒子Z方向的尺寸適度小于晶胞的Z尺寸。具體來說,選擇做NCI分析,進入設置格點界面后,還是選擇模式9,然后輸入以下內容
0,0,1 //盒子原點的X、Y坐標都為0,Z坐標從1 Bohr開始,使得格點不會在晶胞最底層出現
0,0,10.85 //讓盒子的前兩個邊長等同于晶胞的前兩個平移矢量長度,而令盒子的Z尺寸比晶胞Z尺寸小2 Bohr,使得格點不會在晶胞頂端分布(注:晶胞尺寸在Multiwfn載入文件后屏幕上直接顯示了,Z尺寸是12.85 Bohr,減去2就是這里寫的10.85 Bohr)
0.2 //格點間距,同前
用這樣得到的cub文件在VMD里作圖如下所示,可見很理想,只保留了晶胞中間兩層結構間的層間和層內相互作用。
(2)另一種避免顯示與鏡像晶胞間的RDG等值面的方法是在Multiwfn計算時要求不考慮相應方向的周期性,我最推薦這種做法。比如對此例不想考慮Z方向的周期性,就把Multiwfn的settings.ini里的ifdoPBCxyz從原先的1,1,1改成1,1,0,這里0就代表第三個晶胞方向不考慮周期性(由于當前的晶胞第三個方向就是Z方向,所以等于不在Z方向考慮周期性)。這么改過之后照常計算和繪圖即可,格點還是按原先方式設置,結果和上圖完全相同。這種做法不僅方便,省得特意考慮格點分布范圍,而且由于忽略了一個方向的周期性,計算耗時還下降了百分之幾十,一舉兩得。
下面是對當前體系按照3.1.2節的過程繪制的準分子近似的NCI圖的結果,忽略了Z方向周期性。計算耗時極低,只花了1秒就算完了,圖像效果和普通NCI定性一致。
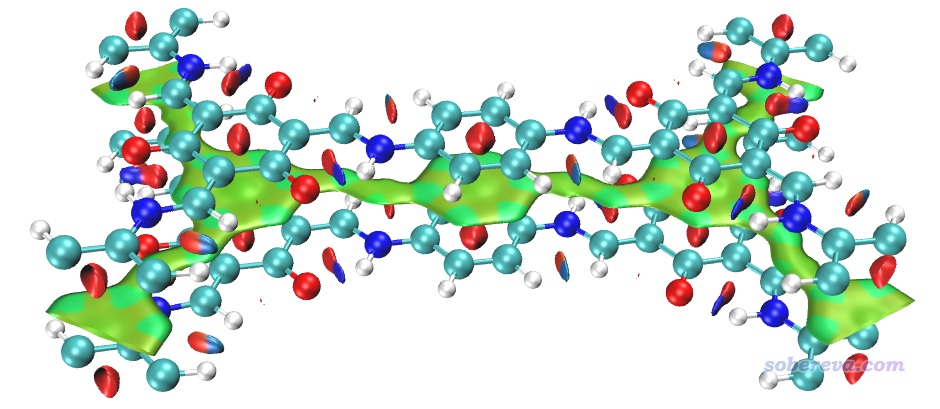
雖然準分子近似版NCI多數情況下和普通NCI定性一致,但是如果你對準確度要求比較高,比如考察氫鍵時想根據等值面藍色的深淺程度判斷強度,那么不要用準分子近似的版本,否則可能得到一些誤導性結論,畢竟用準分子近似相當于沒有考慮原子間相互作用引起的電子轉移、極化效應對NCI分析結果的影響。
3.3 氧化石墨烯與水的相互作用
在《使用Multiwfn非常便利地創建CP2K程序的輸入文件》(http://www.shanxitv.org/587)文中曾使用PBE-D3(BJ)/TZVP-MOLOPT-GTH對氧化石墨烯+一個水分子的表面吸附體系做了幾何優化,計算后得到了opt-1.restart文件,這其實是個CP2K的輸入文件,里面記錄了優化的最后一幀的信息。現在我們對這個結構做個NCI分析。首先基于這個文件創建一個PBE/6-311G**級別下的單點任務文件,用于得到molden文件。
啟動Multiwfn后輸入
opt-1.restart
cp2k
SP.inp
-2 //要求產生molden文件
2 //修改基組
11 //6-311G**
0 //產生輸入文件
用CP2K計算產生的SP.inp,算完后就有了SP-MOS-1_0.molden,然后把SP.inp里的晶胞信息手動寫入到這個molden文件作為[Cell]字段。
啟動Multiwfn,載入SP-MOS-1_0.molden。在計算前我們先進入主功能0看一下這個體系的結構特征,并且選圖形窗口菜單欄的Other settings里的Toggle showing cell frame把晶胞邊框顯示出來,
此時看到的圖像如下所示
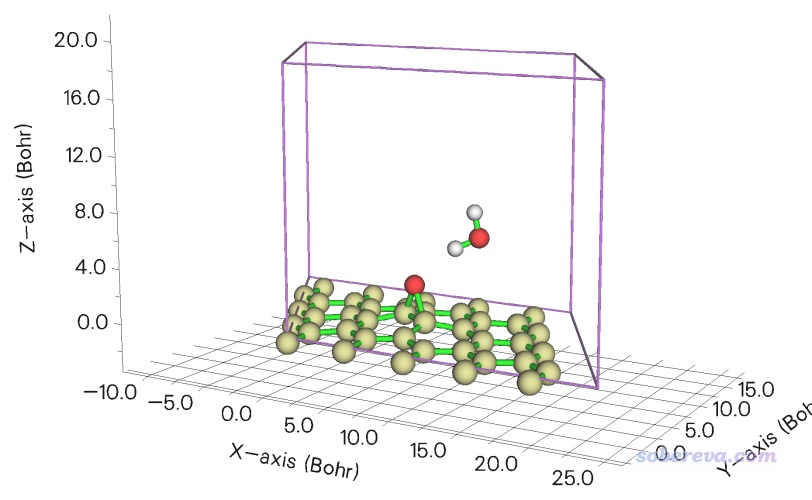
做這個體系的NCI分析時要注意,由于石墨烯板緊貼著盒子最下方,因此如果直接將0,0,0作為盒子原點位置,屆時你會看到石墨烯里六元環中的位阻區域的RDG等值面被截斷,不好看。為避免這個問題,一個做法是用Multiwfn的主功能300的主功能7里的選項1先把整個體系往Z的正方向挪比如2 Bohr(約1埃),另一個辦法是定義格點的時候,讓盒子原點Z位置適當小于0,比如-2.0 Bohr。另外,當前體系水分子上方有大量的真空區,為了節約時間,可以讓Z方向盒子尺寸明顯小于晶胞的Z尺寸(設成其一半就足夠包圍感興趣的弱相互作用區域,可以節約一倍的計算時間)。下面計算時會考慮這兩點。
在當前圖形窗口的右上角點擊RETURN關閉窗口,然后輸入
20 //弱相互作用圖形化分析
1 //NCI分析
9 //借助晶胞信息定義格點
0,0,-2 //盒子原點位置(Bohr)
0,0,9 //當前晶胞Z方向尺寸是18.897 Bohr,令盒子Z尺寸取其一半,其它方向和晶胞尺寸相同
0.15 //格點間距
在筆者36核機子上13秒算完。算完后用選項3導出cub文件,和3.1.1節一樣在VMD里結合RDGfill2.vmd腳本繪制NCI圖,如下所示。為了讓大家了解當前盒子范圍,即格點數據計算的區域,在Graphics - Representation界面里我把對應Isosurface的那個representation里的Show下拉框從默認的Isosurface改為了Box+Isosurface。
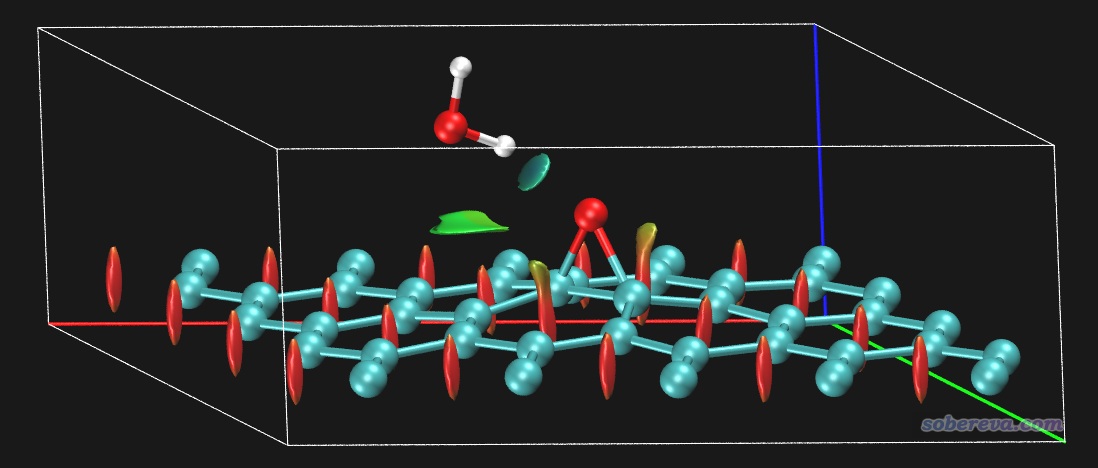
從上圖可見,石墨烯每個環中央的位阻區域被清楚地通過紅色等值面展現了出來。水與氧橋之間有一塊淡藍色圓片型等值面,體現出氫鍵的存在,但顏色不算很深,說明不算很強的氫鍵。在水分子與碳的接觸區域還有一塊綠色的等值面,體現出水和氧化石墨烯之間還有明顯的范德華作用區域。上圖白色方框展現的是盒子范圍,可見盒子取得比較恰當,既沒有截斷任何等值面,也沒有明顯的浪費。由于當前體系與Z方向的鏡像距離比較遠,不會與其相互作用導致出現額外的RDG等值面,此例就沒有刻意把settings.ini里的ifdoPBCxyz像前例一樣改成1,1,0,但如果這么改一下的話倒是可以通過忽略Z方向周期性節約一些耗時。
3.4 C60晶體
此例考察一下C60富勒烯分子晶體中的弱相互作用,本文文件包里C60.cif是此體系的實驗結構。這個體系的晶胞里原子數很多,有240個,若用普通NCI分析耗時會較高,而且此體系主要是pi-pi堆積作用,這種作用靠準分子近似的NCI就可以合理地展現出來,所以此例就只用準分子近似的NCI了。
啟動Multiwfn,然后輸入
C60.cif
20
2 //準分子近似的NCI
9
[回車]
[回車]
0.15 //讓圖像較平滑,用較小的格點間距
計算完后導出cub文件并用RDGfill_pro.vmd在VMD里作圖,如下所示
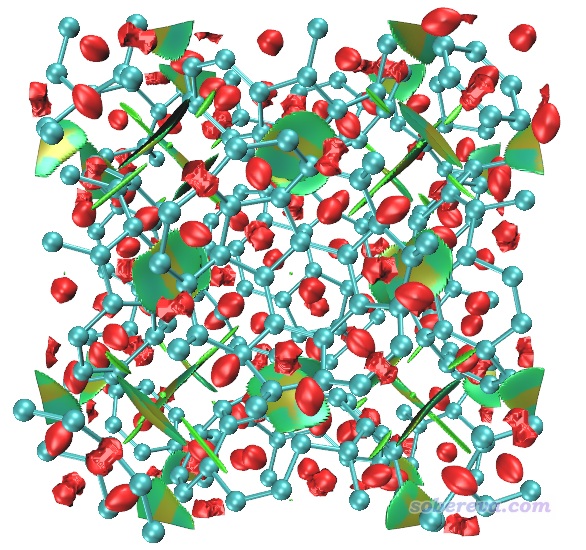
這個圖像還不是很令人滿意,主要是紅色的表現位阻區域的等值面太多了,弄得圖像很凌亂,不便于觀察富勒烯之間的相互作用。為解決此問題,一方面可以用后文說的IGM,另一方面可以把sign(λ2)rho數值明顯大于0的區域手動屏蔽掉。具體做法是在Multiwfn做NCI分析的后處理菜單里選-1先把RDG vs sign(λ2)rho的散點圖繪制出來,如下所示
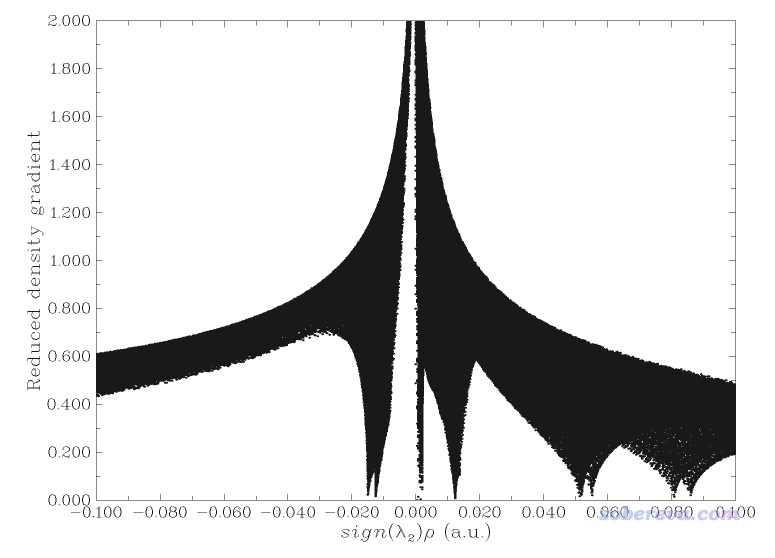
由圖可見在sign(λ2)rho>=0.05的區域有很多戳到底的spike,它們是導致分子內位阻作用的等值面出現的原因。為了屏蔽掉它們,在Multiwfn后處理菜單中選-2 Set RDG value where value of sign(λ2)rho is within a certain range,輸入0.02,999,然后輸入100。此時sign(λ2)rho數值大于0.02的區域的格點的RDG值就都被設為一個很大的數值100了,如果再重新繪制散點圖會看到下圖,可見sign(λ2)rho>0.02區域的spike都已經沒了。
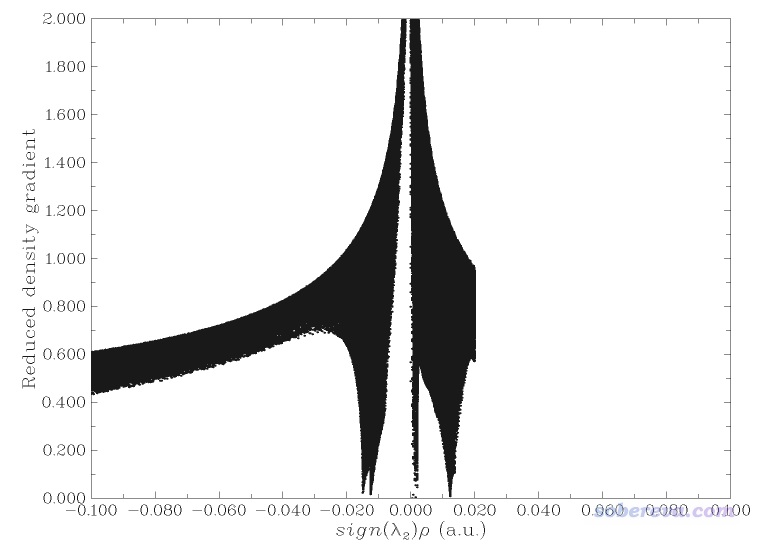
現在重新選擇導出cub文件,然后再次用RDGfill_pro.vmd在VMD里作圖。并且在VMD里把+X的鏡像也顯示出來便于考察。然后在VMD文本窗口里輸入
color Name C tan
color change rgb tan 0.700000 0.560000 0.360000
這會把碳的顏色改成黃褐色,因為原本的青色與等值面顏色的對比度不夠強烈。再在Graphics - Representation里把分子結構顯示方式改為Licorice,并把Bond Radius改為0.1,之后在VMD Main窗口里選Display - Orthographic改為正交視角。此時的圖像如下
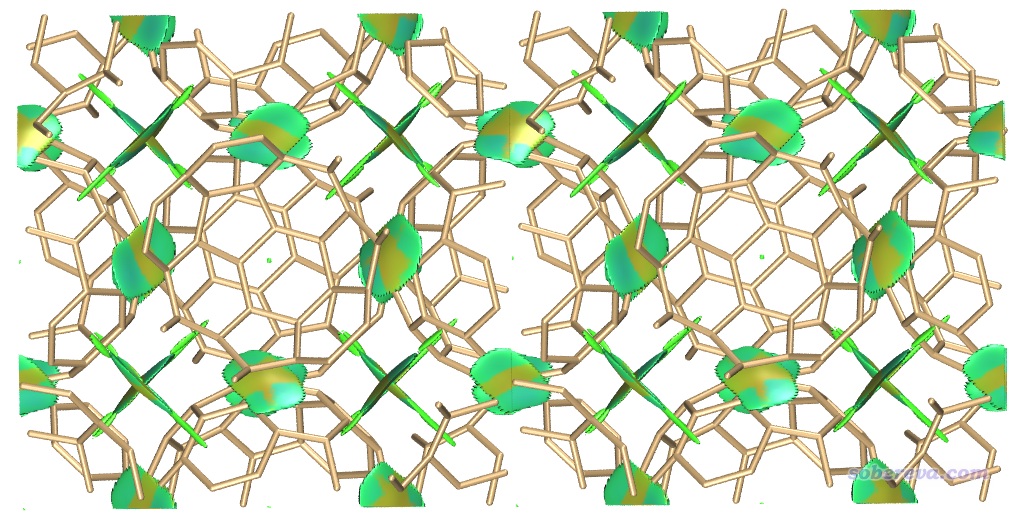
可見當前的等值面清楚地把每個富勒烯與相鄰富勒烯之間的pi-pi堆積展現了出來。肯定有人注意到,上圖中央的富勒烯中間有些C-C鍵是斷開的,而且圖像中央的RDG等值面中間有個黑色的細縫。這是因為VMD沒法識別和顯示當前晶胞里的原子與相鄰鏡像晶胞里的原子間的鍵,而且等值面在跨盒子的地方不可避免地會顯示出個縫隙。如果想避免這倆問題讓圖像更完美,就只能在Multiwfn做這個準分子近似的NCI分析前,先用主功能300的子功能7的選項19把體系構造成2*1*1的超胞,然后再照常做NCI分析。此時計算耗時會明顯增高,cub文件大小也會增加一倍。
3.5 Ih晶型的冰
此例用普通NCI方法考察Ih晶型的冰中的弱相互作用。Ih是常壓下不是特別低溫的情況時最穩定的冰的晶型,也是日常生活中我們接觸到的冰的晶型。大家有興趣也可以類似地考察其它晶型的冰中的弱相互作用。
先產生優化冰晶胞中氫位置的CP2K輸入文件。啟動Multiwfn,載入本文文件包里的H2O-Ice-Ih.cif,然后輸入
cp2k
opt.inp
-2 //產生molden文件
2 //選擇基組
11 //6-311G**
3 //開啟色散校正
2 //DFT-D3(BJ)
-1 //設置任務類型
3 //優化結構但不優化晶胞
9 //設置凍結
optH //只優化氫
0 產生輸入文件
現在用CP2K進行計算,很快就算完了。把晶胞信息手動寫入對應優化好的結構的opt-MOS-1_5.molden文件里作為[Cell]字段。由于冰當中的氫鍵作用相對較強,為了避免氫鍵對應的RDG等值面被截斷,把RDG_maxrho設為0.07。然后照常做NCI分析。由于當前晶胞不大,用很小格點間距要算的格點數也不很多,因此為了圖像效果盡量精甚細膩,此例用了0.13 Bohr這樣很小的格點間距。
照常使用RDGfill2.vmd腳本在VMD里對Multiwfn做NCI分析后導出的cub文件作圖。為了讓冰當中的相互作用顯得比較完整,在Graphics - Representation界面里把顯示分子結構和顯示等值面的representation都設成+X、+Y、-Y、+Z、-Z鏡像都顯示,并且設置霧化效果使得遠處的物體被一定程度遮掩而避免擾亂視線。具體來說就是確保VMD Main的Display里的Depth cueing已經打開了,然后在Display - Display settings里把Cue Mode設成Linear,并把Cue Start和Cue End分別設為1.5和3.0。此時看到的圖像如下。
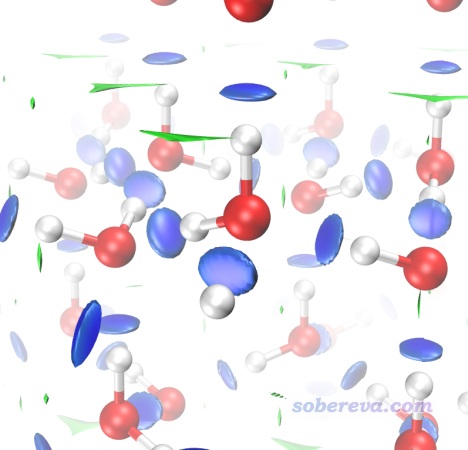
上圖非常清楚地展現出在冰當中,每個水與周圍四個水形成了氫鍵,而且RDG等值面非常藍,體現出氫鍵強度較大。零星的綠色等值面體現出冰當中還有一些主要算是范德華作用的區域,但可以忽略不計。
3.6 NaCl表面吸附CO
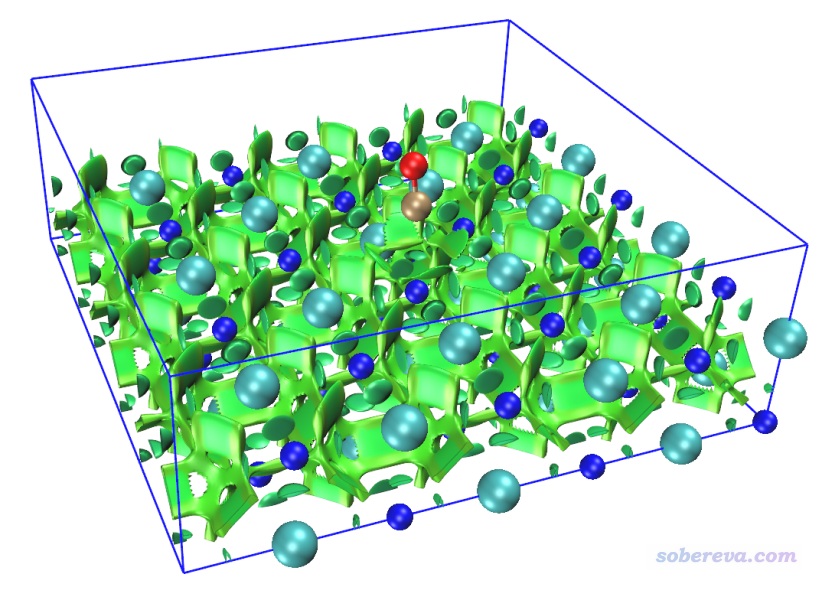
本例繪制NaCl表面吸附一氧化碳的NCI圖。根據諸多文章的研究,最穩定的吸附結構應當是一氧化碳的碳沖著Na+,此例也優化的是這種結構。用的是兩層厚度3*3的氯化鈉板,底層原子凍結,上層和CO都在優化過程中自由弛豫。先通過PBE-D3(BJ)/DZVP-MOLOPT-SR-GTH優化結構,然后PBE/6-31G*算單點得到波函數文件。此模型可以用GaussView很容易地搭建,保存成gjf并載入后通過Multiwfn很容易地就可以創建相應任務的CP2K輸入文件,相關過程這里就不累述了。單點計算后得到的SP-MOS-1_0.molden在本文文件包里提供了。之后對此體系做普通NCI分析,用0.15 Bohr格點間距,圖像如下所示。此體系計算耗時很高,為了節約時間,應恰當設置盒子Z方向尺寸,避免讓格點分布在根本沒原子出現的真空區。
為了看得清楚,俯視圖和側視圖也在下面給出
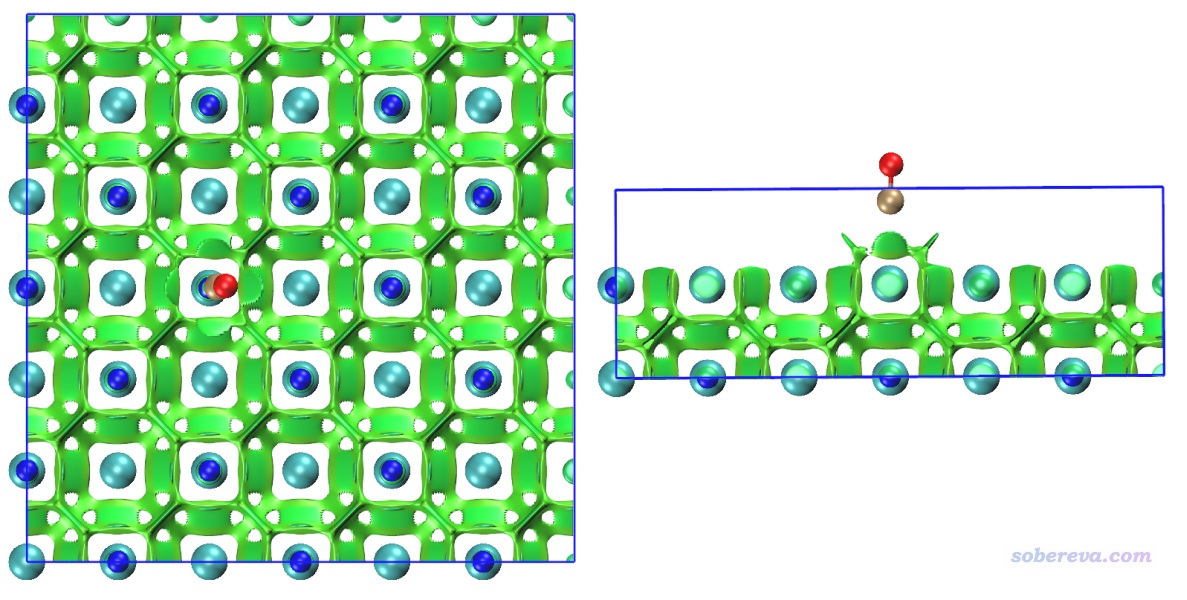
圖中青色圓球是Cl-,藍球是Na+,褐球是碳,紅球是氧,藍色方框展現的是計算的格點數據的分布范圍。從此圖可以清楚地看到一氧化碳的碳與Na+有明顯的相互作用,與旁邊的四個Cl-也有明顯作用,并且Na+和Cl-之間也有顯著相互作用。Na+帶顯著正電,它與碳的鍵軸末端的孤對電子明顯會形成顯著的靜電主導的吸引作用(文獻中的吸附能約為20 kJ/mol,不算小了),且Na+和Cl-之間毫無疑問是挺強的離子鍵,為何RDG等值面全都是綠色而不是藍色?這是因為鈉的半徑較大,而且價層孤立狀態下就只有一個電子,在當前體系中價電子就更少了,故在它與其它原子形成相互作用的區域必定電子密度(rho)比較低,所以sign(λ2)rho數量級很小,故著色是綠色。記住切勿盲目用RDG等值面顏色衡量作用強度,要正確理解NCI方法的本質和局限性。
上面的圖有個明顯的不足之處就是Na+和Cl-之間作用的等值面嚴重妨礙了觀看氯化鈉和一氧化碳之間的相互作用。既可以使用后文所述的IGM方法避免此問題,也可以使用Multiwfn的格點數據處理功能(主功能13)中的子功能14解決,它可以用來將NaCl板與一氧化碳交疊區域以外的區域的格點的RDG設成一個特定值,比如100,從而達到屏蔽NaCl內部相互作用的目的。下面就來這么屏蔽一下。NCI分析后導出的func2.cub是RDG的格點數據文件,將之載入Multiwfn,然后輸入
13 //主功能13
14 //設置兩個片段交疊區域以外區域格點的數值
1.3 //原子球半徑設為范德華半徑乘以1.3(片段占據的區域是片段內所有原子球的疊加)
100 //交疊區域以外的格點數值設為100
2 //手動輸入兩個片段里的原子序號
1-72 //片段1的原子序號,即NaCl板中的原子
73,74 //片段2的原子序號,即CO的原子
0 //將當前的格點數據導出為cub文件
[按回車] //格點數據導出為當前目錄下的func2.cub
現在,用新得到的func2.cub替換原先的func2.cub,再用RDGfill2.vmd腳本作圖,得到如下圖像,可見非常理想,確實NaCl內部的等值面都被屏蔽掉了。為了效果更好,作圖時筆者還設了景深霧化,并且把RDG等值面數值從默認的0.5稍微設大到了0.6,這樣等值面更膨脹一些,邊緣鋸齒看起來也更小一點。
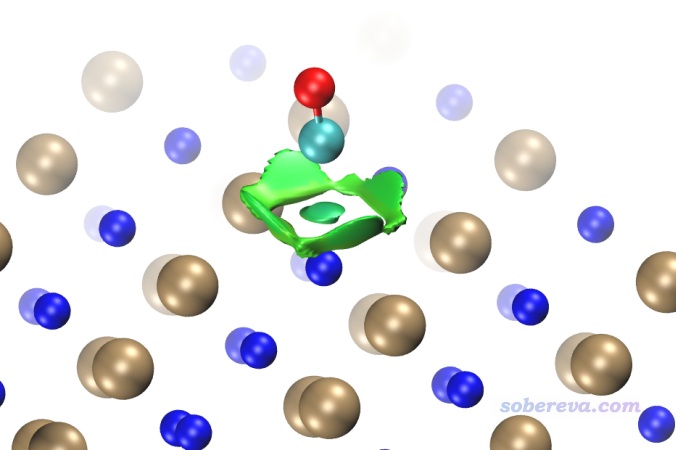
順帶一提,還可以在VMD中使用焦距虛化效果,如下所示,這樣層次更清楚。也可以通過把原子球調大來提升層次感。
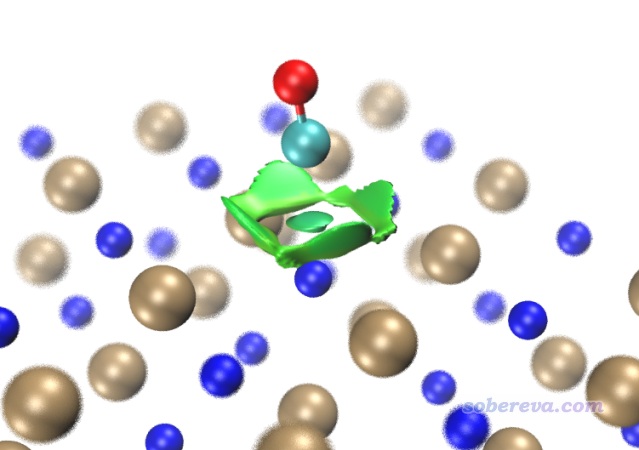
像上面這樣只需要對一小部分區域作NCI圖的情況,在Multiwfn里設置計算的格點范圍的時候可以只讓盒子框住感興趣的區域(即感興趣的等值面可能出現的區域),耗時能節約非常多。這通過恰當設置盒子原點和邊長即可實現,也可以在格點設置界面里通過選項10 Set box of grid data visually using a GUI window在圖形窗口里直觀設置盒子的位置和尺寸,正如此文2.6節所示的情況:《使用量子化學程序基于簇模型計算金屬表面吸附問題》(http://www.shanxitv.org/540)。
4 周期性體系的IGM分析
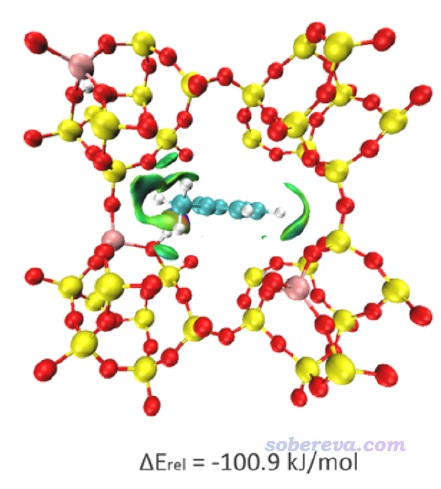
IGM分析只需要原子坐標。如果體系雖然是周期性的,但是被考察的區域與晶胞邊界有一定距離,那么完全可以按照《通過獨立梯度模型(IGM)考察分子間弱相互作用》(http://www.shanxitv.org/407)所述的當成孤立體系考察。比如Mater. Today Commun., 26, 102028 (2021)一文中,在筆者的建議下作者對他們研究的MOF吸附芳香化合物的問題做了IGM分析,如下所示,可見很好地展現出了MOF對分子的吸附時主要的作用區域。像這種情況就當成孤立體系計算做IGM計算就可以,考慮周期性的話耗時會明顯更高。
但也有一些情況IGM等值面要涉及到體系邊緣,這種情況就必須當成周期性做IGM分析了,否則盒子邊緣處的等值面就不正常了,比如3.2節的COF體系就是如此。這里我們就用IGM方法對COF這個體系做一下分析,如果沒讀過《通過獨立梯度模型(IGM)考察分子間弱相互作用》一文的話請仔細閱讀,基本知識本文不再累述。為了不顯示出中心晶胞和相鄰晶胞的相互作用對應的IGM等值面,還是把settings.ini里的ifdoPBCxyz設成1,1,0忽略掉Z方向的周期性。
啟動Multiwfn,然后輸入
COF_opt-1.restart //本文文件包里優化COF中氫原子位置任務產生的記錄最終結構的restart文件
20 //圖形化分析弱相互作用
10 //IGM
2 //定義兩個片段
1-72 //第一個片段的原子序號,即兩層COF其中一層中的原子
73-144 //第二個片段的原子序號,即另一層COF中的原子
9 //借助晶胞信息設置格點
[按回車] //用0,0,0作為盒子原點
[按回車] //用晶胞邊長作為盒子邊長
0.4 //格點間距。IGM等值面不容易像RDG的那么容易有鋸齒,所以可以用明顯大得多的格點間距節約時間
本身IGM分析就很快,而且格點間距又大,在普通4核機子上不到半分鐘就算完了。之后選3導出格點數據,在當前目錄下就有了dg_inter.cub、dg_intra.cub、dg.cub和sl2r.cub,將它們都挪到VMD目錄下。并且把Multiwfn目錄下的examples文件夾中的IGM繪圖腳本IGM_inter.vmd和IGM_intra.vmd都復制到VMD目錄下。
啟動VMD,然后在命令行窗口輸入source IGM_inter.vmd,這會繪制出IGM方法中定義的sign(λ2)rho著色的δg_inter函數的等值面。但是直接顯示出來的等值面非常窄,不能充分展現出兩層MOF間的pi-pi堆積,這是因為默認的δg_inter等值面數值偏大。因此進入Graphics - Representation界面,把Isovalue改成更小的0.005,此時圖像如下所示。可以看到此圖非常清楚地展現出了層間pi-pi堆積作用,而且片段內的相互作用不像NCI那樣被顯示出來,同時等值面很平滑,可見Multiwfn做IGM分析又簡單又快效果又非常好。IGM分析還有一個好處是可以把各個原子的貢獻給出來,并可以由此給原子球著色凸顯較重要的原子,詳見前述的《通過獨立梯度模型(IGM)考察分子間弱相互作用》。
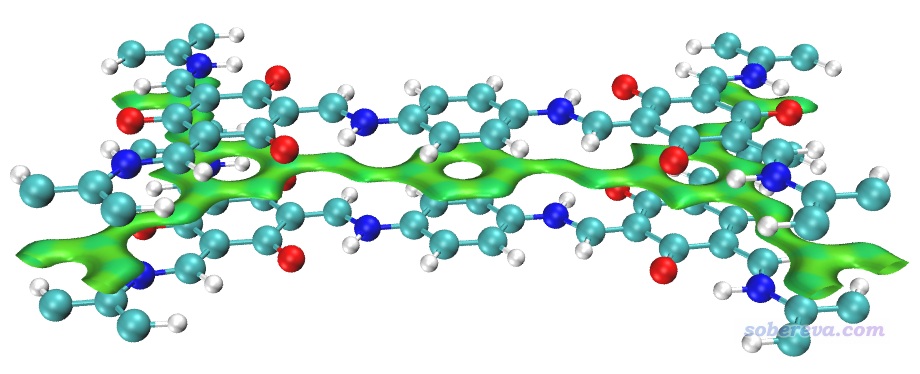
如果在VMD里輸入source IGM_intra.vmd,可以把COF層內的相互作用,包括化學鍵作用和內氫鍵都展現出來(展現后者需要調小isovalue)。由于這不是此例我們感興趣的,就不說了。
下面對本文3.6節考察過的NaCl吸附CO的體系做IGM分析,將NaCl和CO分別作為兩個片段,操作方式同上。下圖左側和右側對應0.005和0.003的δg_inter等值面。
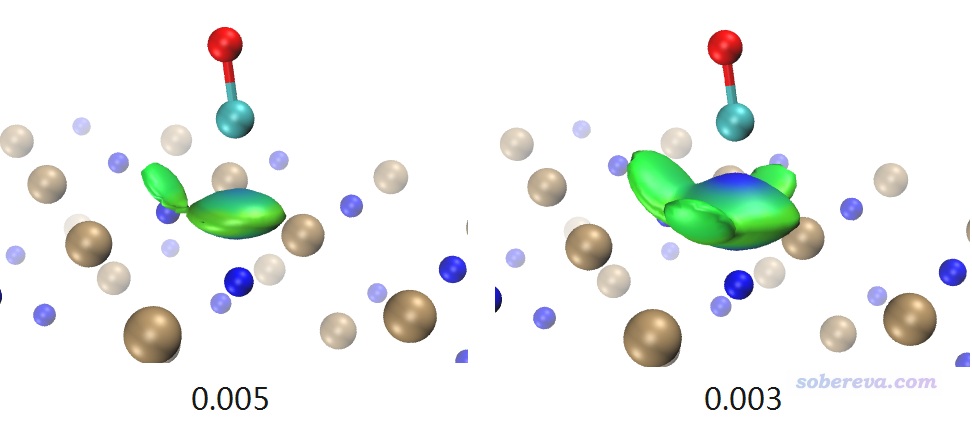
δg_inter數值取得越小,其等值面就可以把越弱的相互作用展現出來。如上所示,在0.005等值面的時候,C和Na+的相互作用區域很明顯,但C和周圍Cl-的范德華作用對應的等值面只顯示出了CO稍微歪過去方向的那一小塊。而將等值面數值降到0.003后,更多相互作用區域被等值面展現了出來,并且此時C和Na+接觸的那部分等值面中央顏色已經很藍了。為什么此時顏色這么藍而不像RDG等值面那樣是綠色的?這是因為此時的δg_inter等值面特別肥厚,已經延展到了比較靠近C的部分,此處電子密度已經不是很小了,sign(λ2)rho可以達到負零點零幾了,因此對應的著色明顯發藍。
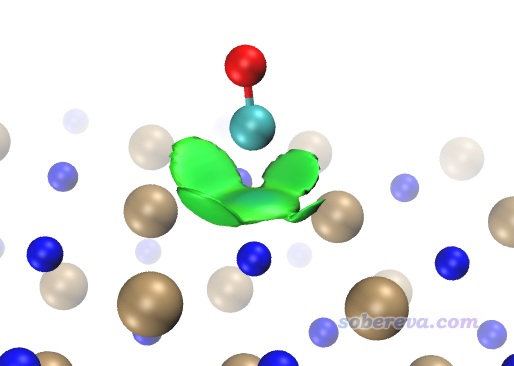
IGM等值面比較肥厚是IGM方法的十分明顯的弊端,而且上面的顏色往往還會誤導研究者,在筆者2022年提出的IGMH方法中被完美解決,而且IGMH比IGM原理更嚴格。讀者務必看《使用Multiwfn做IGMH分析非常清晰直觀地展現化學體系中的相互作用》(http://www.shanxitv.org/621),里面有IGMH非常全面的介紹和與IGM的對比。在Multiwfn中的使用上,IGMH和IGM的區別僅僅在于主功能20里應選擇11而非10,并且需要載入和NCI分析一樣的含有波函數信息的輸入文件。IGMH由于是基于波函數的分析,故耗時顯著高于IGM,而且和NCI分析一樣格點間距不宜太大以避免鋸齒。對當前體系,為節約耗時,建議設置格點時讓盒子僅框住感興趣的作用區域,并且把ifdoPBCxyz設為0,0,0當孤立體系處理(因為CO與NaCl作用的位置離盒子邊緣很遠,這么做不會有不良影響)。基于IGMH方法繪制的δg_inter為0.002的等值面圖如下所示,可見等值面和RDG的等值面很相似,都比較薄,而且和IGM一樣都完全避免了NaCl板內的等值面妨礙考察CO的吸附,圖像效果極度理想。我建議凡是能得到波函數的情況,都應當用IGMH代替IGM。
5 總結
本文詳細介紹了如何使用Multiwfn基于CP2K第一性原理程序產生的波函數做NCI分析,還介紹了怎么對周期性體系做IGM分析,還對筆者提出的比IGM強得多的IGMH分析也做了簡要示例。Multiwfn靈活方便的創建CP2K輸入文件的功能,結合Multiwfn非常簡單易用的NCI/IRI/IGM/IGMH分析功能,真是使得固體和表面的弱相互作用圖形化分析異乎尋常地簡單。研究周期性體系中的弱相互作用不要再只是算算結構、算算能量、只靠數據討論了,結合本文介紹的圖形化分析方法可以令文章大為增光添彩、明顯更吸引眼球,令枯燥抽象的分析討論變得直觀生動傳神。
Multiwfn對周期性體系的分析絕不限于本文所介紹的。目前的版本已可以對周期性體系計算原子電荷、計算鍵級、做AIM拓撲分析、計算和繪制上百種實空間函數(如ELF、LOL、IRI、電子密度拉普拉斯、能量密度、自旋密度等等)、做范德華勢分析(《談談范德華勢以及在Multiwfn中的計算、分析和繪制》http://www.shanxitv.org/551),等等。Multiwfn對周期性體系的支持詳情看Multiwfn手冊2.9節的說明。筆者在未來會陸續寫更多的Multiwfn對周期性體系波函數分析的相關教程。讀者也別忘了看《使用IRI方法圖形化考察化學體系中的化學鍵和弱相互作用》(http://www.shanxitv.org/598),其中有IRI研究周期性體系的介紹,比NCI分析能展現更豐富的信息,建議代替NCI。
順帶一提,如果你之前不會用CP2K的話,也完全不用擔心什么。如本文所示,靠Multiwfn可以非常簡單地創建本來特別繁瑣復雜的CP2K輸入文件,而且如前文提到的我的CP2K安裝教程所寫的,用CP2K預編譯版都完全不需要自己編譯,下載就能用。再加上CP2K又快又免費,結合Multiwfn做周期性體系的波函數分析簡直不能更好用。所以即便你以前用的是Quantum ESPRESSO、CASTEP等其它第一性原理程序,也可以毫無壓力地過度到Multiwfn+CP2K的分析組合上。