幫助設定Gaussian輸入文件中優化flag和MM電荷的小工具
近來由于計算需要寫了兩個小程序setopt和setcharge,一個是用來幫助設定Gaussian輸入文件中的優化flag,另一個是幫助設定MM電荷。雖然程序很簡單,但很有用,這里分享下。Windows版及源代碼下載地址:
optflag:optflag 1.0.rar
setcharge:setcharge 1.0.rar1 optflag程序
Gaussian里optimization flag就是原子后面寫0、-1來分別代表優化時允許移動和被凍結。對于ONIOM,設成同樣的負值的原子還可以被當成剛性片段優化。optflag小程序可以將指定的原子的優化flag設為指定值。來看一個例子:
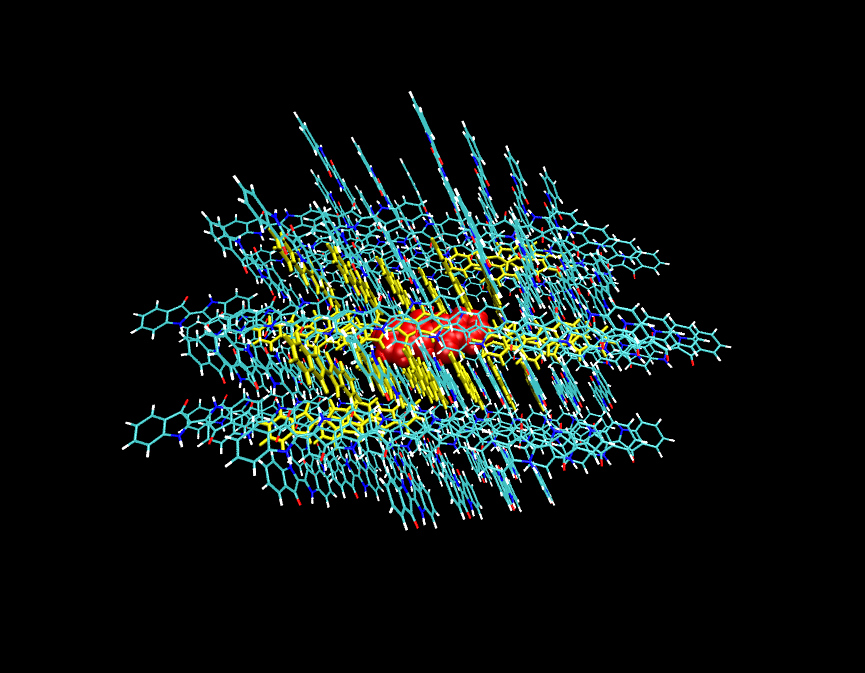
這是個靛藍(indigo)的分子團簇,從晶體結構中截取出來的。pdb文件在此:/usr/uploads/file/20160606/20160606121956_93869.rar
我們想把中間紅色的分子的周圍的一圈分子,即黃色區域的靛藍分子的優化flag設為0,允許它們在優化時調整結構,而將最外圍那些以及中間的紅色靛藍分子設為凍結。
要做到這個目的,我們先用VMD生成原子序號列表。選擇范圍對應于:
中心分子:residue 43
中心分子+相鄰部分:same fragment as within 6 of residue 43
比如要生成中心分子的序號列表,就在VMD控制臺里輸入atomselect top "residue 43",比如提示atomselect0,就再輸入atomselect0 list得到序號列表和atomselect0 num得到原子數。然后寫成索引文件center.txt,將用于作為optflag的輸入,內容如下。
-30
1290 1291 1292 1293 1294 1295 1296 1297 1298 1299 1300 1301 1302 1303 1304 1305 1306 1307 1308 1309 1310 1311 1312 1313 1314 1315 1316 1317 1318 1319
第一行是原子數,之后是原子序號。由于VMD輸出的序號是從0開始的,原子數要寫成負值來指明這一點。以同樣步驟,生成中心+相鄰部分分子的索引文件small.txt。
然后用gview打開.pdb文件,保存成.gjf文件,把原子坐標那部分截出來,并且去掉(pdbname...)那一堆,保存到all.txt里面。
雙擊啟動optflag,輸入
all.txt
4530 //總原子數
0 //當前輸入文件里還沒有優化flag
all //選全部原子
-1 //要設成的優化flag
此時提示在當前目錄產生了new.txt,打開一看,已經相對于all.txt把-1的優化flag加上了。
然后把中心+相鄰部分分子優化flag設為0,啟動optflag輸入
new.txt
4530
1 //輸入文件里已經有優化flag了
small.txt //用來選定中心分子+相鄰部分的索引文件
0 //要設的優化flag
最后一次,將中心分子優化flag設為-1,啟動optflag輸入
new.txt
4530
1
center.txt
-1
好啦,此時得到的new.txt的內容就已經可以拷到Gaussian輸入文件的坐標部分了,優化時就只有中心分子臨近的一圈分子會被優化了。感興趣的話可以把gjf文件拉進gview,進edit-atom groups,選freeze分類,然后高亮或隱藏不同的部分檢查是否設定合理。
2 setcharge程序
Gaussian中用分子力學計算的時候涉及到給原子設定原子電荷,對于體系中包含許多同種分子,比如包含一大堆水分子,或者上面的例子包含一堆indigo,要給自行挨個給同種分子填上原子電荷信息很麻煩。setcharge可以幫助做這個事情。
對于上面的例子,我們要給團簇所有indigo分子設定在indigo孤立狀態下算出的CHELPG電荷,并且假定中間的indigo分子帶+1電荷,因此要給它賦上indigo陽離子狀態下計算的CHELPG電荷。做法如下:
先對indigo在中性和+1狀態下用Multiwfn計算CHELPG原子電荷(參考http://www.shanxitv.org/441),然后分別寫到indigo.txt和indigo+1.txt,第一行是原子數,之后是每個原子的電荷,例如indigo.txt
30
0.099919
0.519588
-0.183248
-0.016362
-0.189175
0.020601
-0.308034
0.418772
-0.659064
-0.562279
0.099919
0.519588
-0.659064
-0.183248
-0.562279
0.418772
-0.016362
-0.308034
-0.189175
0.020601
0.102237
0.100428
0.081598
0.143281
0.431738
0.102237
0.100428
0.081598
0.143281
0.431738
先給所有indigo都賦上中性下indigo的CHELPG電荷,啟動setcharge,輸入
new.txt //這個是上個例子最后得到的new.txt
4530
1
all //給全部原子賦上電荷
indigo.txt //含有原子電荷的文件
原先new.txt里的優化flag還都保留著。當前體系一共4530個原子,indigo.txt里有30個原子的電荷,所以會給1~30、31~60、61~90...4501~4530都依次賦上indigo.txt里的電荷。此時可以看到new.txt里的原子變成了諸如這樣
C---0.308034
即曰C的原子電荷為-0.308034。前兩個橫杠之間是原子類型,這里沒設所以留空。
然后給中心原子賦上+1態的電荷。啟動setcharge,輸入
new.txt //這個是上個例子最后得到的new.txt
4530
1
center.txt //中心分子的索引文件,見上例
indigo+1.txt //含有+1態CHELPG電荷的文件
然后new.txt里的信息就可以拷到gjf文件里用了。