VMD顯示軌跡中粒子的空間密度分布的兩種方法
文/Sobereva
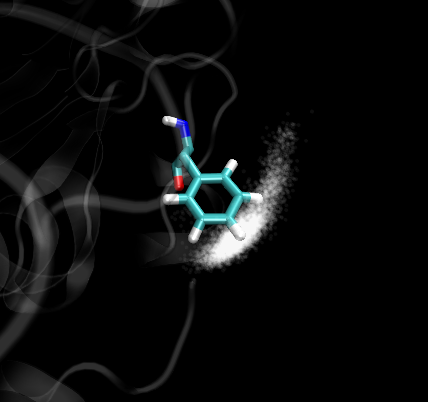
第一種方法是用顏色深淺顯示某原子在不同位置出現的概率,這種方法適合描述少數幾個粒子的密度分布,效果比較柔和。
渲染模式開GLSL,在Materials對話框里把Transparent材質復制到一個新的材質叫a
把a的Ambient設最大,Opacity設0.03,然后在文本控制臺source一下dis.tcl腳本。
運行draw material a
然后比如想看index 444原子在前4444幀內的空間分布狀況,輸入dis 444 4444。如果效果不好,再調a材質的Opacity。
原理簡單來說,就是開了GLSL渲染模式之后所繪的東西可以有透明色,每次出現用一個透明的白色球來表示,這樣的東西疊加次數越多,某處越不透明,顯得顏色越深(圖中的例子就是越白),說明出現概率越大。我的例子是苯丙氨酸苯環末端的H在各個位置的出現幾率,說明不易偏離其穩定位置,越靠邊概率越小。
球半徑0.13是我覺得圖像效果比較好的情況,可以自己調。如果不滿意,就運行draw delete all刪除所繪的一切內容,調完參數再次重復上面步驟運行。
dis.tcl腳本內容:
proc dis {atom fps} {
draw color white
for {set i 0} {$i<$fps} {incr i 1} {
set nowselect [atomselect top "index $atom" frame $i]
set x [$nowselect get {x}]
set y [$nowselect get {y}]
set z [$nowselect get {z}]
draw sphere "$x $y $z" radius 0.13
}
}
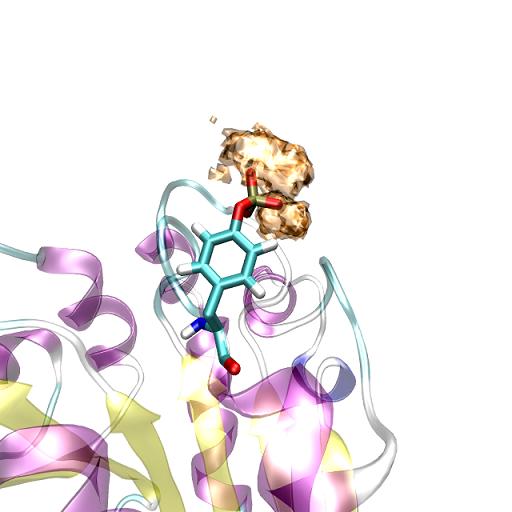
第二種方法是用等值面顯示,這種方法適合描述大量粒子的空間分布。用g_spatial可以計算某個group在空間中每個點的出現幾率。
首先用trjconv -f a-sol.trr -s a-sol.gro -fit rot+trans按照某個東西align一下,比如研究溶劑在蛋白附近分布,fit組就選蛋白,也就是以蛋白不發生整體運動為依據改變整個軌跡中的原子坐標,然后g_spatial -s a-sol.tpr -f a-sol.trr -n index.ndx可以計算某個group在空間中每個點的出現幾率,存為grid.cube。如果說內存分配不夠或者段錯誤,用-nab,設100一般夠了。設bin控制格點寬度,默認是0.05,0.03的時候文件已經很大了。
g_spatial第一次選擇的group決定了空間格點各個值的大小,也即決定了grid.cube的數據部分,得到的等值面圖是什么樣完全取決于第一次選擇的group。第二次選擇group實際上是選擇輸出哪些原子到grid.cube頭部的原子敘述部分,這個完全不影響結果,隨便設,一般設成溶質。比如第一組設的是溶劑,第二組設的是溶質,讀進VMD,用CPK等模式看到的是溶質的結構,用isosurface模式看到的是溶劑出現密度的等值面。兩個模式疊加顯示就是溶劑與溶質的關系。