使用g_densmap得到分子附近二維密度分布圖
Gromacs的g_densmap可以把體系中某個組的出現密度投影到一個平面上,-aver設定一個軸,默認是z,也可以是x和y,投影到的就是垂直于這個軸的面,如z就是xy。-xmin和-xmax設定這個軸上的指定坐標范圍內的粒子被投影到那個平面上。得到.xpm圖。
g_densmap還可以做軸向-徑向密度分布圖。用-rmax定義徑向最大距離,-amax定義軸向最大范圍。需要定義三個組,第一個組的質心和第二個組的質心用來確定軸的方向,這兩個組質心的中間點作為坐標零點,方向是第一個組質心向第二個組質心。第三個組就是求密度的組。得到的xpm的橫坐標是這個軸的不同位置,縱坐標是徑向距離,顏色是密度。也就是這個軸的不同高度位置的垂直于軸的平面的徑向分布函數。
但是有時需要求某些粒子在某個分子周圍分布的二維密度圖,比如這里要得到一個磷酸化酪氨酸附近的鈉離子出現密度,而且苯環與二維圖的平面相平行。粒子的三維空間分布見我曾經發的《VMD顯示軌跡中粒子的空間密度分布的兩種方法》(http://www.shanxitv.org/14)。
和g_spatial一樣,需要先對那個殘基進行fit,但是g_densmap只能求x、y、z方向的投影,并不能自定義投影方向將粒子密度投影到苯環平面上。所以在fit前,需要把初始結構中磷酸化酪氨酸的苯環進行旋轉,使其與某兩個坐標軸組成的平面平行。這一步可以用VMD,選mouse-move-residue,按住shift拖動鼠標左鍵或右鍵旋轉分子,這里讓苯環與yz平面平行,然后save coordinate保存到新的pdb。
tip:旋轉分子的時候可能不好判斷是否與坐標軸平行,因為VMD的坐標軸在左下角不好對照。所以可以自行繪制出xyz坐標軸以便參考。在控制臺運行:
draw color red
draw cylinder { 0 0 0 } { 80 0 0 }
draw cone { 80 0 0 } { 85 0 0 } radius 4 resolution 20
draw color green
draw cylinder { 0 0 0 } { 0 80 0 }
draw cone { 0 80 0 } { 0 85 0 } radius 4 resolution 20
draw color blue
draw cylinder { 0 0 0 } { 0 0 80 }
draw cone { 0 0 80 } { 0 0 85 } radius 4 resolution 20
得到的坐標軸的0點就是真實空間的0點,也可以改在分子附近的位置。
然后用trjconv將軌跡向這個結構fit,選磷酸化酪氨酸組作為參考組。
軌跡處理好后,用VMD估一下合適的xmin、xmax范圍,也就是苯環平面上下幾埃的區域,這里定為z=4.2至4.8nm范圍。運行g_densmap -f A.xtc -s A.pdb -aver x -xmin 4.2 -xmax 4.8。組選擇鈉離子那組。然后再運行一遍這個指令,選磷酸化酪氨酸那組。再把兩個xpm圖用xpm2ps轉為.eps。
鈉離子的密度分布圖:
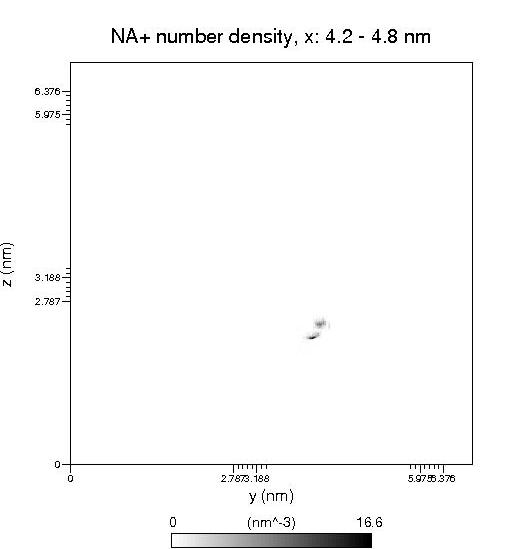
磷酸化酪氨酸的密度分布圖:
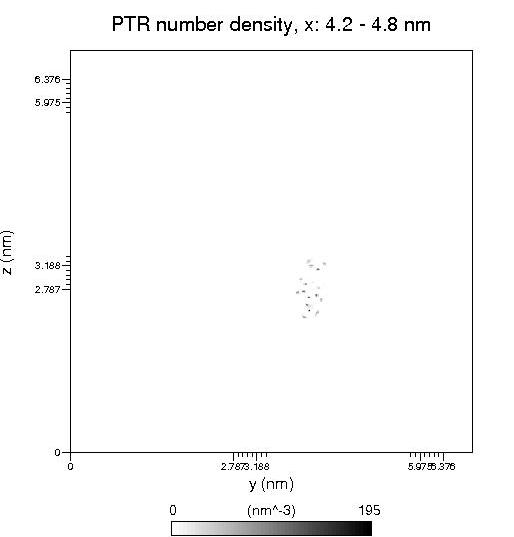
之所以要磷酸化酪氨酸的密度分布圖,是為了定位用,因為還要把磷酸化酪氨酸分子結構畫到鈉離子密度分布圖的對應位置。用photoshop等軟件打開,把這兩個圖疊起來,如圖:
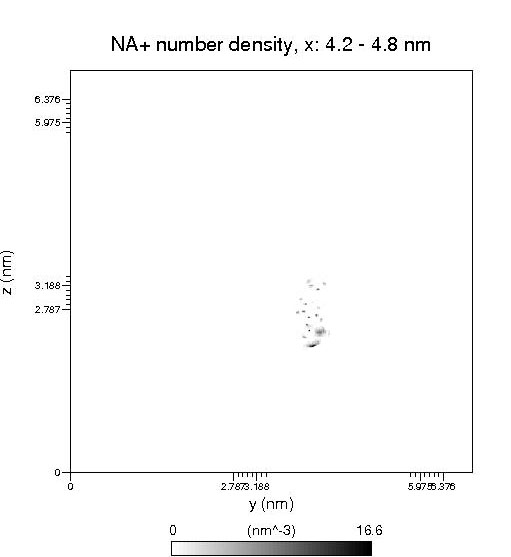
然后自行將磷酸化酪氨酸的結構圖復制到上面即可,圖中可以清楚地看到苯環,把結構圖大小調整合適將苯環重合就行了,很簡單,就不多說了。
這個例子比較糙,沒調坐標軸刻度,也忽略了磷酸化酪氨酸的分子內坐標的變化等問題,總之大概意思就是這樣了,細節請自行研究。