生成混合組分的磷脂雙層膜結構文件的工具genmixmem
生成混合組分的磷脂雙層膜結構文件的工具genmixmem
文/Sobereva@北京科音
First release: 2014-Jul-23 Last update: 2021-Nov-14
1 前言
構建磷脂膜的程序和辦法不少,比如CHARMM-GUI、Packmol等等,但都有這樣或那樣的缺點。比如Packmol構建雙層膜往往要迭代很多次才能收斂,得到的結構往往還有好多空隙。而CHARMM-GUI是在線程序,沒法離線使用,而且支持的磷脂數目有限,碰上一些特殊的磷脂、雙層膜中插入的小分子,CHARMM-GUI就沒法用了,而且還只支持CHARMM力場。
為了能靈活、方便、快速地構建磷脂雙分子膜,筆者專門開發了這個genmixmem程序。只要提供磷脂(或者膽固醇之類小分子)的結構文件,以及一些參數信息,程序就可以瞬間構建出磷脂雙分子膜,尤其適合用于GROMACS中模擬的目的。
genmixmem最新的1.2版下載地址:http://www.shanxitv.org/soft/genmixmem_1.2.zip
其中有預編譯好的Windows下的可執行文件(genmixmem.exe)、Linux下的可執行文件(genmixmem)、源代碼以及示例文件。
如果此程序在你的文章中使用了,請這樣引用:Tian Lu, genmixmem program, http://www.shanxitv.org/245 (accessed month day, year)
PS:筆者之前還寫過《生成磷脂雙層膜結構文件的小工具genmem》(http://www.shanxitv.org/222),但genmem只能用于產生純膜。genmixmem已經可以完全取代genmem的功能。
2 一個例子
此程序使用很簡單。在程序目錄下寫個名為input.txt的輸入文件,然后啟動程序即可。下面直接給出一個例子輸入文件,同時附帶注釋
mixmem.pdb //輸出文件名
5 //磷脂的類型數
64 //每層磷脂的數目
64 //讓磷脂均勻排布在邊長為多大的盒子里(埃)
0 //1=允許讓磷脂繞Z軸隨機旋轉, 0=不允許, -1/-2/-3=在第二層里旋轉45/90/180度
80.5,39.5 //參考原子所在的第一層和第二層的Z坐標位置
0 //是否讓兩層磷脂圖案對稱,0=不要求,1=要求
#
#下面是每個磷脂類型的結構文件,在每層中的數目,以及參考原子是幾號原子
#
pdbinput\DGDG343z.pdb,20,27
pdbinput\LysoPGz.pdb,10,5
pdbinput\MGDG366z.pdb,14,26
pdbinput\PA342z.pdb,15,21
pdbinput\PC364z.pdb,5,24
對于此例,啟動genmixmem后,程序就從指定的那些pdb中讀取磷脂結構,根據指定的每種磷脂的數目和設定的相關參數產生雙分子層結構,輸出到當前目錄下的mixmem.pdb。當前目錄下還出現了mixmem.txt,內容是各個磷脂的殘基名在mixmem.pdb里的出現順序和數目。如果你要用GROMACS做當前體系的模擬,mixmem.txt里的內容應當都拷到拓撲文件的[ molecules ]字段里,這樣就和mixmem.pdb精確對應上了。
此例中,每層的8*8=64個磷脂分子均勻分布在64*64埃的區域里,磷脂都是順著Z方向。每種類型的磷脂的分布是隨機的,每次運行程序給出的排布都會不同,因此可以用來產生大量不同的初始結構。此例在生成膜結構時沒有允許磷脂分子繞著Z軸隨機或以某種方式旋轉。第一層和第二層膜中每個磷脂的參考原子分別要求出現在Z=80.5和39.5埃的平面上,參考原子選的是每個磷脂的丙三醇部分的中央那個碳(由于這個碳通常在磷脂的最關鍵的位置,所以通常用它當參考原子來對磷脂定位,當然也可以取別的原子)。此例用了5種磷脂,想增加或減少類型數的話,在輸入文件末尾增加或刪去磷脂定義,并且修改input.txt的第二行的數字即可。
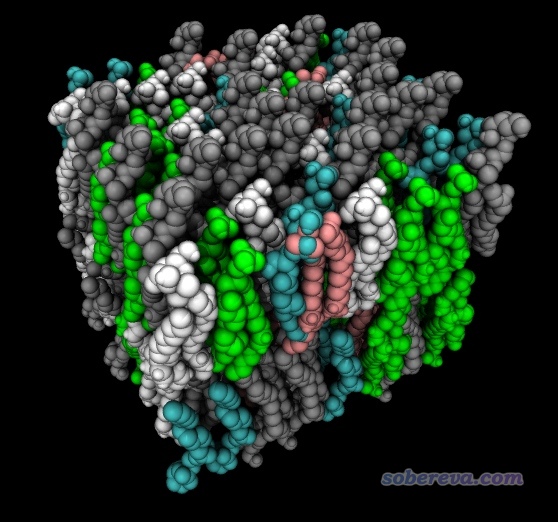
此例得到的結果如下
如果讀者用的是Linux版genmixmem,注意此例末尾5行應當寫成這樣
"pdbinput/DGDG343z.pdb",20,27
"pdbinput/LysoPGz.pdb",10,5
"pdbinput/MGDG366z.pdb",14,26
"pdbinput/PA342z.pdb",15,21
"pdbinput/PC364z.pdb",5,24
3 相關使用細節
輸入的磷脂pdb文件中,磷脂頭部應當沖著Z軸正方向。旋轉分子可以在VMD或GaussView等程序里做,也可以通過GROMACS的editconf命令來做,比如
gmx editconf -f MGDG366.pdb -o MGDG366z.pdb -align -1 0 0 -princ -center 0 0 0
如果之后發現MGDG366z.pdb里的頭部方向恰好反了,就把上面命令里的-1改成1。
磷脂結構文件里的磷脂應當比較直,以避免生成的結構中相互交錯而導致不合理接觸。可以用可視化程序SAMSON(https://samson-connect.net)把磷脂拉直,里面有個twist工具實現這個目的超級方便,筆者錄了一段演示視頻請觀看:http://www.shanxitv.org/attach/245/SAMSON_straighten.mp4。可惜SAMSON目前免費版里面twist工具已經沒法用了,需要花錢買收費版(雖然收費版也可以申請免費試用)。筆者還總結了其它幾種把分子結構拉直的簡便做法,見《利用Gaussian或ORCA程序把分子結構拉直的幾種方法》(http://www.shanxitv.org/594)。
雖然此程序目的是產生磷脂層,但組成并不限于磷脂分子,比如包含膽固醇等其它分子也都可以,只要設置對應的pdb文件即可。
普通的磷脂頭部的面積在65 Angstrom^2左右,因此你可以由此大致估計盒子邊長設多少合適。比如每層64個的話,盒子邊長用sqrt(65*64)=64.5埃往往是個大致合理的數值。
genmixmem并不會像Packmol那樣利用優化算法來讓磷脂分子剛性地旋轉以避免過近的接觸。雖然此程序生成的膜結構中難免有些不合理接觸,但這不是什么問題,因為MD之前做優化就能解決不合理接觸。即便沒法徹底解決掉某些局部區域的高斥力,只要做MD一開始的時候用非常小的步長,比如0.5fs,稍微跑一下也就解決了。
順帶介紹一個筆者做磷脂膜模擬的經驗。如果在磷脂兩端加水之后做MD的過程中發現有水鉆進了磷脂膜的疏水部分,可以給水在Z方向加上限制勢,跑幾百ps之后磷脂分子就恰當地弛豫了,磷脂之間也沒有什么縫隙了。此時去掉限制勢再做常規模擬時,水分子也就沒機會鉆進磷脂膜的疏水區了。
4 關于創建磷脂膜的拓撲文件
在北京科音分子動力學與GROMACS培訓班(http://www.keinsci.com/workshop/KGMX_content.html)講生物膜模擬的部分我非常詳細講怎么基于genmixmem構建的膜結構做分子動力學模擬,包括拓撲文件的準備過程。鑒于也經常有genmixmem的用戶在網上問我怎么得到做GROMACS模擬對應的拓撲文件,我這里就很簡單地說一下。
專門為生物膜提出的力場有很多,如kukol(JCTC, 5, 615)、Poger(JCTC, 6, 325、JCC, 31, 1117)、54A8_v1(JCTC, 15, 5175)、Slipids(http://www.fos.su.se/~sasha/SLipids/Downloads.html)、Lipid17(https://github.com/xiki-tempula/gmx_lipid17.ff),等等。比如說你做膜模擬打算用kukol力場,在JCTC, 5, 615 (2009) DOI: 10.1021/ct8003468一文的補充材料里可以得到它支持的DPPC、DMPC、L-POPG、D-POPG、POPC磷脂的itp文件,以及預平衡好的各種膜的pdb結構文件。為了確保磷脂分子的拓撲文件里的原子順序和膜結構里相應磷脂分子的原子順序相一致(這是GROMACS能正常模擬所要求的),建議從力場作者預平衡好的整個膜的pdb文件里摳出來各類磷脂部分保存成磷脂分子的pdb文件(還要按照前述做法拉直),然后用它們基于genmixmem構建磷脂的結構文件。膜體系的top文件自己按照GROMACS拓撲文件一般規則手寫就行了,比如構建的膜里面有POPC和DMPC兩類磷脂,就要在里面恰當位置寫上#include "popc_53a6.itp"和#include "dmpc_53a6.itp",這倆itp是kukol膜力場文章補充材料里給的POPC和DMPC的itp文件(如果它們不在當前目錄下的話要寫實際的路徑)。不同磷脂力場兼容不同的主力場,會用到里面的原子類型,比如kukol磷脂力場與G53A6力場兼容,它用到了G53A6里的原子類型,所以顯然要把G53A6的主top文件forcefield.top也include。genmixmem運行完之后會在mixmem.txt里給出各個磷脂出現順序,它與產生的磷脂膜結構文件里磷脂的出現順序對應,要將它們拷到top文件的[ molecules ]里面。
下面一個典型的top文件例子,有64個DMPC和64個POPC,在genmixmem產生的磷脂膜結構的基礎上還用gmx solvate加了SPC水(必須手動去掉出現在磷脂膜疏水區的水,最后剩下4455個),所以[molecules]最后有SOL 4455,而且還include了spc.itp。[ molecules ]里的POPC和DMPC分別對應popc_53a6.itp和dmpc_53a6.itp里定義的磷脂分子名字。
#include "gromos53a6.ff/forcefield.itp"
#include "gromos53a6.ff/spc.itp"
#include "popc_53a6.itp"
#include "dmpc_53a6.itp"
[ system ]
64 DMPC + 64 POPC in water
[ molecules ]
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
POPC 1
POPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
DMPC 1
DMPC 1
POPC 1
POPC 1
POPC 1
SOL 4455