Sob談生物膜體系的搭建
注:本文已經沒什么意義了,用筆者后來開發的genmixmem工具來構建磷脂膜方便得多效果也明顯更好,見http://www.shanxitv.org/245。而本文的把蛋白嵌入膜的方法也不再推薦,建議使用GROMACS直接支持的membed方法,在北京科音(http://www.keinsci.com)舉辦的分子動力學與GROMACS培訓班里會講解和演示
Sob談生物膜體系的搭建
文/Sobereva @北京科音 寫于約2008年
生物膜體系似乎很多人覺得比較難建,都得從網上下載別人現成的似的,甚至弄到一些就感覺如獲至寶,其實生物膜體系僅僅結合chem3D和packmol就可以隨心所欲地搭建。mdbbs上有個人問如何得到“把9個DPPS脂質分子嵌入到121個DPPC分子得到的膜,并且置于在5476個水分子中”,此文中我們就來建立這個體系。
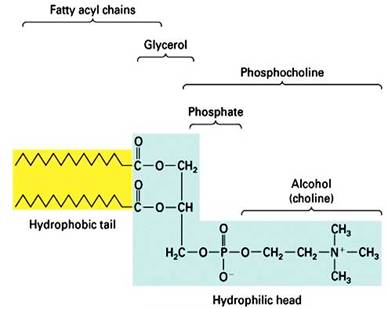
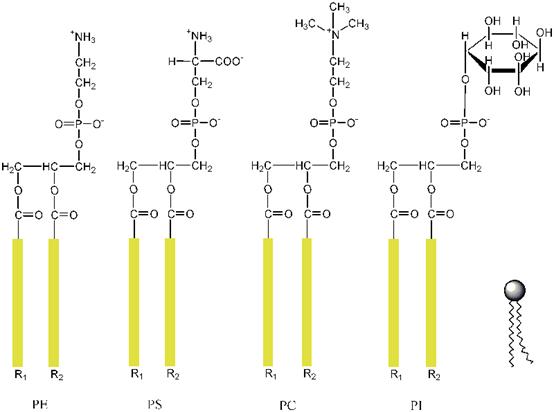
我先說說膜的基本結構。膜脂是膜的基本骨架,膜脂主要包括磷脂、糖脂和膽固醇三種類型,而磷脂占整個膜脂的50%以上。磷脂主要包括脂肪酸鏈、甘油、磷酸酯三個部分,種類很多,在不同生物不同細胞中分布不同。
磷脂經常用四個字母表示,比如DPPS、DOPE之類的。磷酸酯的酯部分的不同,決定了后兩個字母,比如PS就是磷脂酰絲氨酸(phosphatidyl serine),PE就是磷脂酰乙醇胺(phosphatidyl
ethanolamine,舊稱腦磷脂),等等。而前兩個字母和它們的脂肪酸鏈有關。在Wiki上有一個不錯的縮寫和完整寫法的對照,我打印成了pdf發在這里。
/usr/uploads/file/20150602/20150602215525_49469.pdf
為什么要強調它們的名字呢?,最方便的方法就是直接在ChemBio3D里面輸入!CCDC里面雖然有很多小分子晶體結構,但里面磷脂分子很少,所以CCDC沒什么用,得自己搭。
這里先制作DPPC分子的結構文件,查找那個縮寫大全.pdf,得知DPPC的完整寫法是1,2-Dipalmitoyl-sn-Glycero-3-phosphocholine。我這里用的是ChemBioOffice 2008 Ultra 11.0.1。啟動ChemBio3D之后,點擊工具欄上的A按鈕(圖1),在空白處點一下,輸入1,2-Dipalmitoyl-sn-Glycero-3-phosphocholine,回車,得到了DPPC(圖2)
圖1
圖2
但是發現兩個脂肪鏈相距太遠,搭磷脂層的時候肯定會互相碰上,所以我們要把兩個脂肪鏈并到一起。這里要用到ChemBio3D自帶的MM2力場優化功能。點擊Single Bond按鈕,把兩個脂肪鏈的碳原子每隔兩個碳鏈接一次(圖3)。
然后點擊MM2優化按鈕(圖4)。
優化之后,兩條鏈并起來了(圖5)
然后再把鍵打開,選擇一個原子后,按住shift再點與之相連的另一條鏈的原子,兩個原子呈黃色后,按Ctrl+B,鍵就斷開了(圖6) 。如果沒斷開而且還自動加了氫,應當在File-Model setting-Mode building里面把rectify去掉。
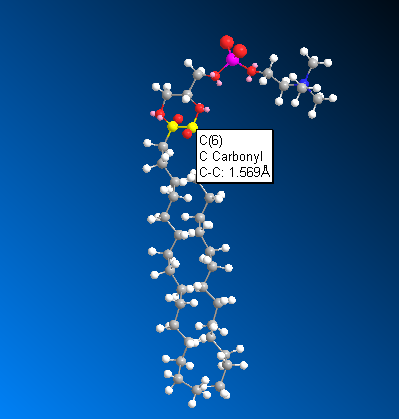
然后再點MM2按鈕來優化。
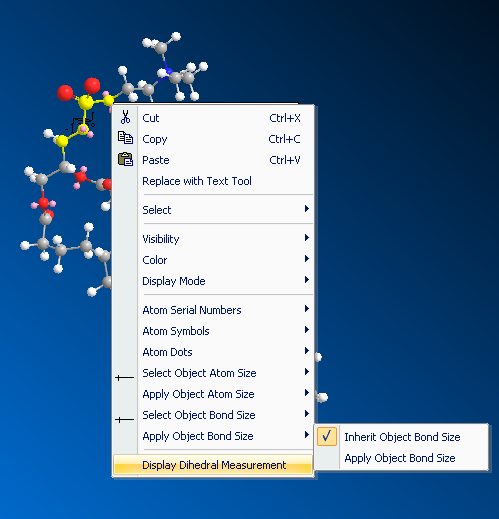
但是還有個問題,就是磷酸膽堿那個部分比較歪,搭建膜的時候時候也會礙事,所以應當擺正。找到比較關鍵的二面角,按住shift連續點擊四個原子,在某個原子上點右鍵選Display Dihedral Measurement(圖7)
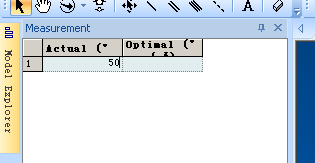
在左側出現二面角數值,改成50(圖8)
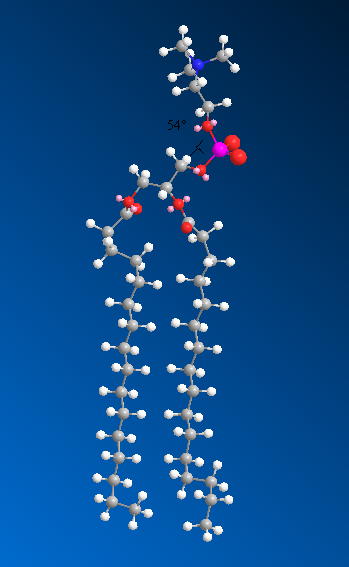
最終再優化一下,得到圖9的樣子,保存為a.mol2。(最好別保存為pdb,似乎chemBio3D有bug,尤其是MM2優化之后保存為pdb容易莫名其妙出錯退出)
a.mol2里面的原子名是很荒涼的,最好走一遍prodrg賦上有意義的名字,而且生成的.itp還有用。把a.mol2的內容復制到prodrg2.5 beta里面(http://davapc1.bioch.dundee.ac.uk/cgi-bin/prodrg_beta),注意EM選NO,否則兩條脂肪酸鏈往往又跑開了。
運行完之后,點擊"Download everything as a gzipped tarfile or as a zip archive (read 00README in the archive)."里面的zip archive,把其中DRGFIN.PDB(加了全部氫)、DRGGMX.PDB(加了極化氫和芳香環上的氫,不含其它氫,與DRGGMX.ITP相對應)、DRGGMX.ITP(gmx會用到的top文件)提取出來。
用同樣的方法得到DPPS的pdb和itp。
DPPC和DPPS的結構已經準備好了,現在用packmol建立磷脂雙層體系模型
在http://www.ime.unicamp.br/~martinez/packmol下載packmol和Input example文件。
解壓,直接輸入make就可以編譯,調用的是gfortran。如果要安裝并行版本,輸入make
parallel。編譯得到packmol可執行文件。
把DPPS.pdb和DPPC.pdb都放到packmol可執行文件所在目錄,并把example里面的water.pdb也拷到這個文件夾。
編寫packmol的輸入文件bi.inp,內容如下(不要把注釋也寫進去):
tolerance 2.0 //原子間最近距離不得小于2.0filetype pdb
output bilayer.pdb //輸出bilayer.pdb
structure water.pdb //設置第一層水
number 2738 //水分子數量
inside box 0. 0. 0. 72. 72. 25. //定義2738個水分子所在的范圍,水分子會取向隨意地分布在這個范圍內,六個數即x/y/z-min,x/y/z/-max。這些坐標憑直覺就能確定,我這里不用圖來說明了。
end structure
structure water.pdb //設置第二層水
number 2738
inside box 0. 0. 75. 72. 72. 100.
end structure
structure DPPC.pdb //設置DPPC分子
number 61
inside box 0. 0. 20. 70. 70. 50. //DPPC所在范圍
atoms 28 //DPPC的第28號原子,也就是膽堿的N
below plane 0. 0. 1. 25. //前三個數定義一個平面,這里定義的就是Z方向,必須小于25埃的位置
end atoms //結束第28號原子的說明
atoms 1 //脂肪鏈最末端的C
over plane 0. 0. 1. 48 //必須Z>48,這樣限制了DPPC兩端原子的坐標范圍,所有DPPC分子的方向就基本定了,但應當留有一些任意旋轉的余地,應該事先量一下這兩個原子間的距離,在DPPC中是25.84,大于48-25=23,所以是可行的。如果想讓DPPC取向再亂一些,定義的兩個平面之間的距離應該再縮短。
end atoms
end structure
structure DPPC.pdb
number 60
inside box 0. 0. 50. 70. 70. 80.
atoms 1
below plane 0. 0. 1. 52.
end atoms
atoms 28
over plane 0. 0. 1. 75
end atoms
end structure
structure DPPS.pdb
number 5
inside box 0. 0. 20. 70. 70. 50. //水、DPPC、DPPS定義的范圍有交集,沒有關系,packmol不會使它們之間位置產生沖突,會自動排列讓它們避開,DPPS和DPPC定義同樣的范圍,會均勻地混合在一起。
atoms 27
below plane 0. 0. 1. 25.
end atoms
atoms 1
over plane 0. 0. 1. 48.
end atoms
end structure
structure DPPS.pdb
number 4
inside box 0. 0. 50. 70. 70. 80.
atoms 1
below plane 0. 0. 1. 52.
end atoms
atoms 27
over plane 0. 0. 1. 75.
end atoms
end structure
現在執行./packmol < bi.inp。packmol其實收斂得挺慢的,和體系、設定都有關系,不要故意去等,先干別的,等什么時候算完了再看結果,結果總是不錯的。往往是超過了迭代次數上限自動停止,會輸出帶FORCE后綴的.pdb,實際上結果已經很好了。最終得到的結果(圖10):
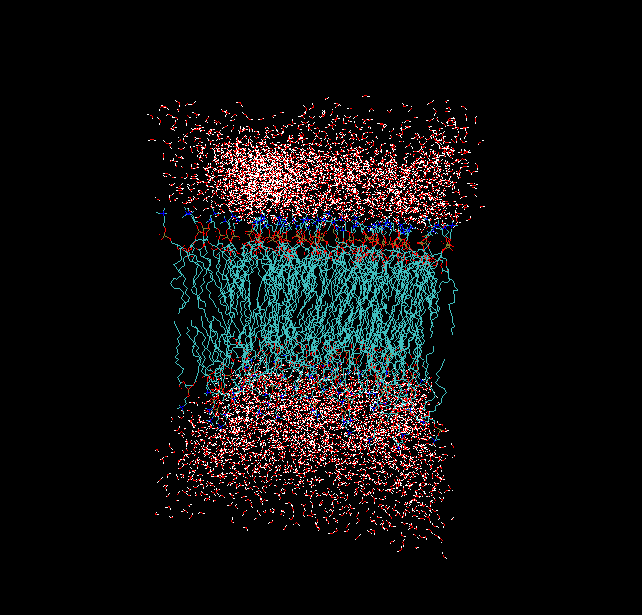
如果DPPS和DPPC混合得不是太均勻,不滿意的話,可以再次運行。也可以用FIXED關鍵字來特別指定DPPS所在位置,見packmol的user guide。
體系分子越多定義越復雜packmol收斂越慢,做一個新的體系之前,最好先每個部分單獨試驗,很快就能得到結果,比如先不考慮水,只生成DPPS和DPPC部分看看能否得到正常結果。加上水之后收斂頗慢,加水的工序可以用gmx的genbox來做,然后用我寫的grostat的反選功能和保留完整殘基功能來去掉兩個磷脂層疏水區的水。
其實還有各種各樣的方法都可以建這樣的體系,比如MS里面晶胞平移、leap里面平移、復制循環使用,gmx里面genconf等等,但都有種種缺點而且麻煩。上述過程是最佳途徑。
得到的bilayer.pdb可能殘基名、殘基序號等等不如意,解決方法很多也很簡單,不多說了。因為構建膜時候就是用的prodrg輸出的pdb,所以和prodrg輸出的.itp順序都是一樣的,用gmx跑的時候可以直接include prodrg得到的itp,這就避免了用網上下載的膜體系和prodrg輸出的itp原子順序不對的問題(可以用itp2rtp解決,見相應readme)。也可以用我寫的itp2rtp,由itp生成用于.rtp的數據,再用pdb2gmx把膜讀進去。
下面討論一下把膜蛋白嵌入磷脂膜的方法
有一些專門的軟件可以做效果也好,但這里我想說說簡單直觀的方法。
用我寫的grostat可以做,確定好膜蛋白在膜中預計的坐標和它跨膜區域所占面積,啟動grostat,讀入膜的gro(可以用editconf直接在pdb和gro格式之間來回轉換),選擇反轉柱形選擇模式,輸入柱的位置、半徑,選擇保留完整殘基,然后再用VMD把膜蛋白拉入膜的圓柱形的洞里面,但是這樣還是有點不夠直觀,跨膜部分的形狀不一定就是標準圓洞形,故適用范圍有限。不如下面的方法。
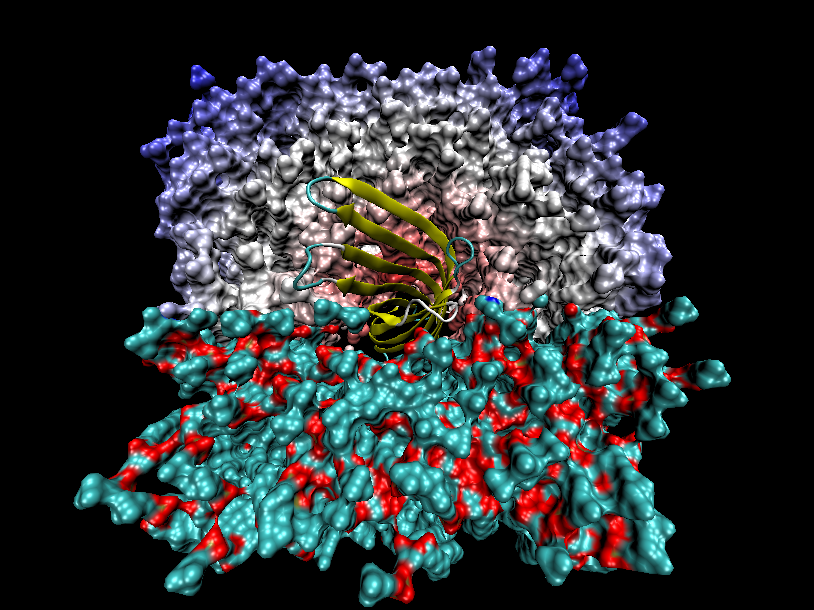
這是我發現的一個簡單的直接用VMD在磷脂膜上挖洞放膜蛋白的方法。這里隨便從RCSB弄一個膜蛋白舉例,首先通過上面的方法得到一個雙層DPPC,面積稍微大一些100*100埃^2,每層150個DPPC。然后用VMD同時打開這個膜蛋白和磷脂層的pdb,看看膜蛋白是否和磷脂層有重合,如果重合的話,按數字7,然后拖動膜蛋白使之不與磷脂層接觸。然后用save coordinate功能把膜蛋白保存成新的pdb。然后把這個新的pdb和磷脂層的pdb直接用文本編輯器合并到一起比如叫a.pdb,用gmx的editconf -f a.pdb -o a2.pdb,這樣a2.pdb里面的殘基號都是順著的,應當手動檢查一下看看殘基編號有無問題。
然后用VMD打開a2.pdb,建立一個層用new cartoon顯示蛋白,另一個層用line顯示,所選的內容是all not same resid as (within 1.5 of protein),然后按數字7打開fragment平移模式,按住shift拖動膜蛋白調好方向,然后用鼠標在不同視角下直接拖拉膜蛋白,使膜蛋白嵌入磷脂膜當中合適位置,就做好了。然后還是save coordinate功能,選擇all not same resid as (exwithin 1.5 of protein),保存為新的pdb就可以了。圖中是此法弄好的膜蛋白嵌入磷脂膜的結構。當然這個方法很粗糙,蛋白對周圍磷脂的擠壓等問題也沒考慮,但是十分簡便易行。
上面的選擇范圍all not same resid as (within 1.5 of
protein)代表的是與蛋白中任意原子距離小于1.5埃的原子,它所在的整個殘基都不顯示。注意within也包括了of后面即蛋白質自己的原子,所以這句話,不僅說明與膜蛋白相近的磷脂不以line來顯示,膜蛋白本身也不以line來顯示,免得看起來亂。而保存時候選擇范圍必須把within改成exwithin,是因為exwithin相對于within,不包含of后面內容中的原子,拆開來說,
exwithin 1.5 of protein 選擇的是:距離蛋白質任何原子1.5埃以內的非蛋白質自身的原子
same resid as (exwithin 1.5 of protein) 選擇的是:距離蛋白質任何原子1.5埃以內的非蛋白質自身的原子所在的整個殘基
all not same resid as (exwithin 1.5 of protein) 選擇的是:全部原子除了距離蛋白質任何原子1.5埃以內的非蛋白質自身的原子所在的整個殘基。換句話簡單來說選擇的就是:蛋白質自身、距離蛋白質1.5埃以外的DPPC。離蛋白質較近的DPPC被剔掉了。結果如圖11所示,顯示的是切面圖。
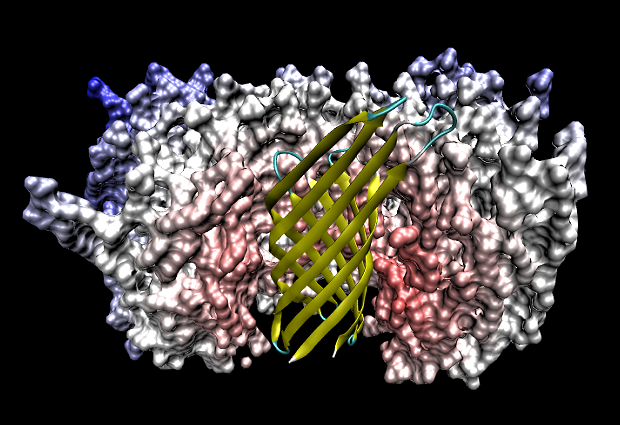
在這里介紹的最后一種方法是使用packmol,在構建的時候就直接把膜蛋白與磷脂混在一起而不是在建好了膜之后把膜蛋白塞進去。然而packmol基于文本操作不夠直觀,而且速度比較慢,但是效果更好,磷脂與膜蛋白帖得比較緊。
首先打開膜蛋白,比如a.pdb,按數字鍵7,按住shift拖動蛋白使蛋白位置比較好,一般讓跨膜的軸垂直于Z軸,即與磷脂膜是垂直的。然后save coordinate到1.pdb,放到packmol可執行文件所在目錄。
然后寫一個in.inp然后 用packmol執行
tolerance 2.0 //使得膜蛋白與磷脂膜分子之間距離不會小于2埃
filetype pdb
output bilayer.pdb
structure 1.pdb
number 1
center //選擇幾何中心
fixed 50. 50. 60. 0. 0. 0. //說明將膜蛋白的幾何中心固定到x/y/z為50,50,60的位置,后面三個數字控制膜蛋白的旋轉角度,因為剛才已經用VMD修改了膜蛋白的朝向,與Z軸平行了(即垂直于磷脂膜),所以這里不用讓它旋轉。
end structure
structure dppc.pdb
number 150
inside box 0. 0. 20. 100. 100. 50.
atoms 28
below plane 0. 0. 1. 28.
end atoms
atoms 1
over plane 0. 0. 1. 48
end atoms
end structure
structure dppc.pdb
number 150
inside box 0. 0. 50. 100. 100. 80.
atoms 1
below plane 0. 0. 1. 52.
end atoms
atoms 28
over plane 0. 0. 1. 72
end atoms
end structure
然后得到的結果如圖12所示。