談談體數據3:用Multiwfn、Gaussview、Molekel、VMD觀看龍蝦、盆景、骨盆、大腦
談談體數據3:用Multiwfn、Gaussview、Molekel、VMD觀看龍蝦、盆景、骨盆、大腦
文/Sobereva @北京科音 2012-Feb-22
原本打算標題定為《使用計算化學可視化軟件觀看與化學無關的體數據》,但感覺太刻板吸引不了眼球,就改成當前這個標題了。這篇帖子是《談談體數據》系列文章之三,比較有趣味,將介紹如何用一些常見的計算化學可視化軟件,包括GaussView、VMD、molekel,以及Multiwfn(http://www.shanxitv.org/multiwfn),通過等值面的方式觀看亂七八糟的與化學毫無關系的體數據。這有助于了解顯示體數據的操作方法,也有助于了解體數據的意義和價值。此文將涉及到《談談體數據》系列的前兩篇文章的知識,建議先看一看,第一篇是《談談體數據1:介紹體數據》,見http://www.shanxitv.org/127。第二篇是《談談體數據2:體數據格式轉換工具vodaconv的使用及原理》,見http://www.shanxitv.org/128。
1 用Multiwfn觀看龍蝦
從Multiwfn2.3開始,Multiwfn可以讀入cube文件,可以直接用于顯示cube文件的等值面。這里用它來觀看盆景的體數據。
用Multiwfn觀看龍蝦就必須得到相應的體數據文件。在Volrenapp的老版體數據庫http://www.gris.uni-tuebingen.de/edu/areas/scivis/volren/datasets/datasets.html里面找到龍蝦圖案并點擊,得到lobster.raw.gz。此數據庫里的體數據都是8bit精度,這個是龍蝦的CT掃描得到的體數據,從旁邊的信息得知這個龍蝦的體數據的點數是301x324x56,格子比例是1:1:1.4。
在這里下載vodaconv程序:/usr/uploads/file/20150611/20150611014131_45262.rar。將lobster.raw.gz解壓為lobster.raw,并拷到vodaconv所在目錄下。啟動vodaconv.exe,依次輸入
lobster.raw //文件名
301,324,56 //各方向的點數
0.1,0.1,0.14 //各方向格子間距(bohr)。絕對數值無所謂,只要比例是1:1:1.4就行了。設為0.1數量級是因為這種情況下體數據的各方向總長度將會在一般的分子尺度內,方便觀看
[直接敲回車] //raw格式體數據沒有定義原點,這一步直接敲回車的話將vodaconv將自動把原點設定在體數據中心
1 //表明這是8bit整數數據
2 //不對數據做scale,保持數據原樣
lobster.cub //輸出的文件名,根據后綴名程序會知道要輸出為Gaussian型cube文件
lobster.raw僅有5.2MB,而轉換后卻有69.5MB。這是因為.raw是二進制文件,一個8bit數據點只占一個字節。而cube文件記錄的是字符數據(也有二進制型cube文件,但不被普遍支持),每個數據要用13個字符來表達,即13字節,因此轉換后大小約是之前的13倍。
將Multiwfn目錄下的settings.ini里的aug3D參數從默認的6改成16并保存,這樣方可讓坐標軸足夠長,而令龍蝦能顯示完整。
啟動Multiwfn,輸入lobster.cub文件的路徑,然后選功能0,等一會兒就會出現圖像。由于這個體數據的格點數遠比一般的分子的體數據要多,因此會顯示得頗慢。圖像第一次出現時是在默認的0.05的isovalue下做的圖,并沒顯現出龍蝦。在isosurface value文本框里輸入50,敲回車,等一會兒就會看到龍蝦了:
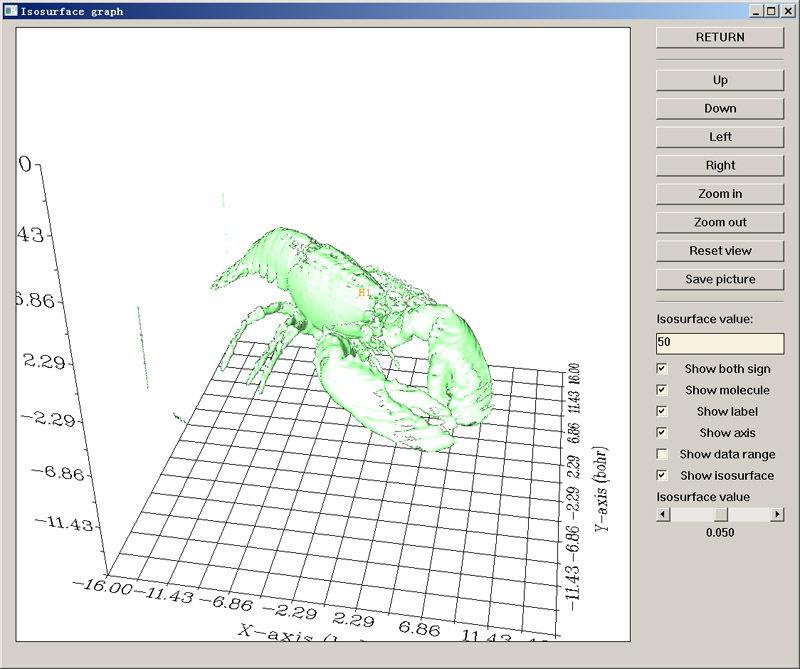
從raw轉換為cube文件時為了兼容性考慮,會自動在原點加入一個氫原子,因此會有一個氫原子標簽,可以點一下Show label復選框來關掉之。
之所以將isovalue設為50,是因為將它設為這個值的時候對于這個體數據來說圖像效果較好。8bit整數范圍在0~255,需要在這個范圍內反復設定isovalue并作圖才能找出一個最合適的isovalue值。一開始作圖時由于默認的0.05的isovalue過小,龍蝦被CT掃描時產生的數據噪音或者周圍空氣產生的信號所掩蓋了所以看不出來。
Multiwfn提供顯示等值面的功能只是為了提供一個方便,對于這樣格點數很多的情況顯示等值面的速度比Molekel、VMD低得多。
2 用Gaussview觀看盆景
還是在剛才的數據庫中,點擊盆景圖案下載盆景的CT掃描的體數據文件bonsai.raw.gz,用和上一節完全一樣的步驟,通過vodaconv將之轉化成bonsai.cub。注意各方向點數應輸入256,256,256,輸入間距的時候直接敲回車就行了,代表用默認的0.1,0.1,0.1,因為這個體數據文件是1:1:1的格點間距。
啟動Gaussview,進入打開文件界面,文件類型選Cube files,選擇bonsai.cub。選Results-Surfaces/Contours,此時會看到已經有一個cube文件被載入了。將Density設為50(這也即isovalue的值),然后點Surface Actions-New surface,耐心等一陣子,然后將視角拉遠,就會看到盆景了(為了清楚,背景改為了白色。通過在圖上點右鍵-View-Hydrogens可以將額外加入的氫屏蔽掉):
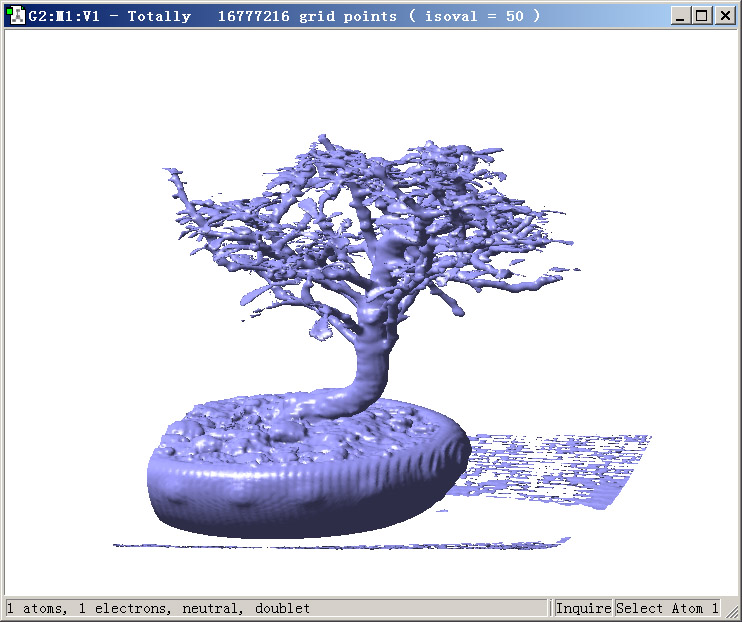
Gaussview在顯示大型體數據的等值面的時候也很慢,因此沒什么實際應用價值。顯示幾百萬個點的體數據的等值面的速度尚可,而當前體數據則有256^3=1600萬個點。對于精度更高的體數據,比如512^3=1.34億個點的情況,用Gaussview顯示等值面就別想了。
3 用Molekel顯示骨盆和腹部動脈支架
本節用Molekel顯示骨盆和腹部動脈支架。在Volrenapp的新版數據庫http://www.gris.uni-tuebingen.de/edu/areas/scivis/volren/datasets/new.html里面下載Stented Abdominal Aorta 16Bits對應的stent16.raw.zip,解壓成stent16.raw。
這個數據庫中的16bit整數記錄的體數據都是Little Endian順序,因此應當使用vodaconv壓縮包里的vodaconv_LE.exe來做轉換,而不是使用用Big Endian順序的vodaconv.exe做轉換。關于這一點我在《體數據格式轉換工具vodaconv的使用及原理》里已經詳細說明了。
啟動vodaconv_LE.exe,依次輸入
stent16.raw
512,512,174
0.8398,0.8398,3.2
[回車]
2 //讀入的raw文件的數據類型是16bit整數
2 //不scale數據
stent16.cube //后綴名這回寫成.cube而非.cub,實際上里面的格式都一樣,只是Molekel在讀入cube文件時必須要求后綴是.cube
啟動Molekel,這里用的是5.4.0.8版。在其中打開stent16.cube,耐心等待讀取完畢,然后選Surfaces-Grid data,在Value里填1300。這個體數據很大,如果想全精度地顯示出來,那么應保證有1.5GB空余內存;如果想損失一些精度,以節省內存且讓顯示速度加快,那么可以將Step Multiplier設為2或者更大。
最后點Generate按鈕。此時會看到程序左下角生成等值面的進程,待生成完畢后,將視角拉遠,就會看到骨架了:
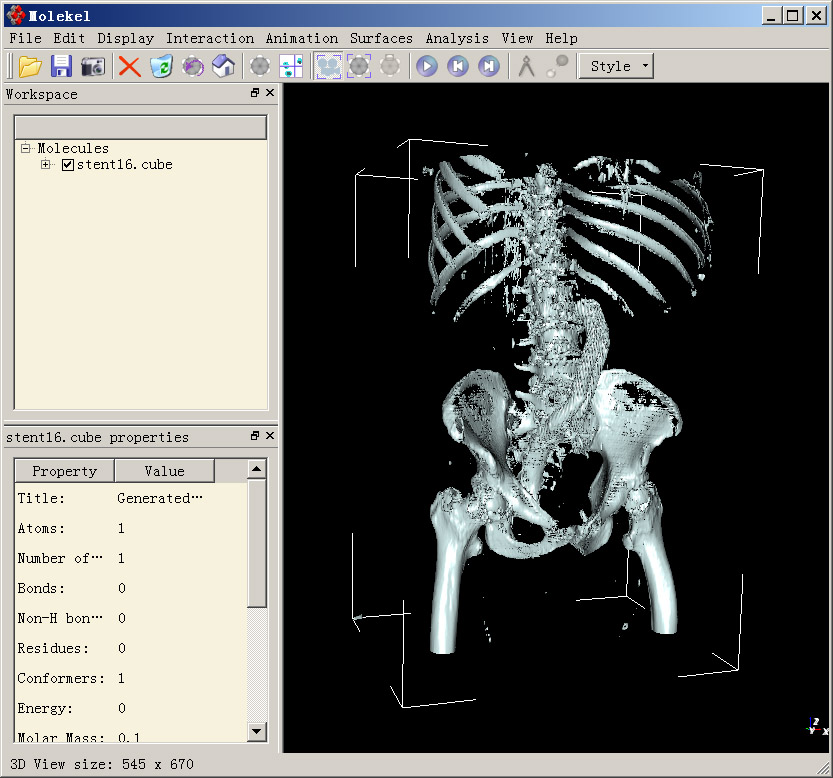
你會發現肚子中央有兩條網狀的物體,那個就是腹部動脈支架。
如果將Value值設小一些,比如設為700,將會使X射線更容易穿透的區域也被顯示出來,因此你將會看到身體表層的結構,以及做掃描時后背貼著的背板的結構:
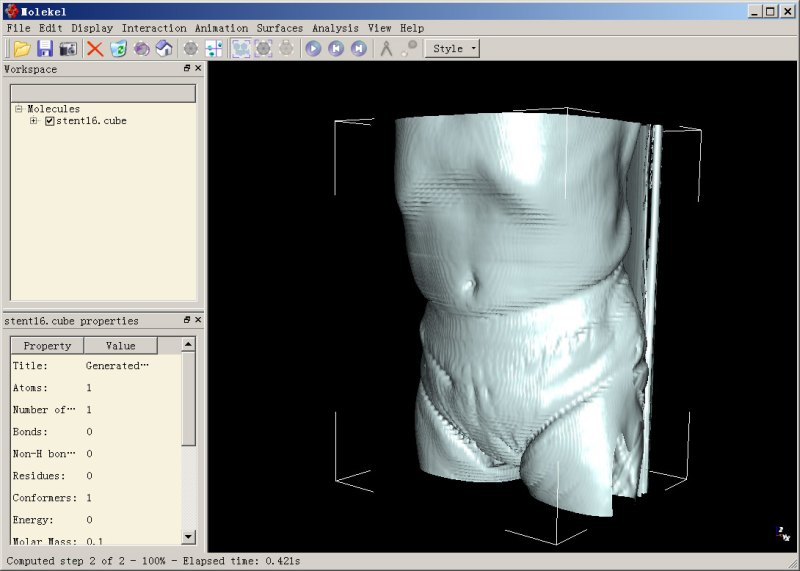
Molekel的bug略多,生成等值面的過程中可能會崩潰、或者顯示不出來等值面,不要覺得奇怪。整體來說,還是下面用的VMD在顯示大型體數據的等值面時最穩健,速度也快,而且更為靈活。
4 使用VMD顯示大腦結構
本例將使用VMD顯示DTI(彌散張量成像)方法產生的大腦結構的體數據。DTI是通過外加磁場,根據測定的腦實質中水彌散的各向異性對大腦進行定量成像的方法。
進入http://www9.informatik.uni-erlangen.de/External/vollib/,在網頁中搜索4977B2FE,點擊對應的Downlaod按鈕得到DTI-B0.pvm。pvm格式是V3體數據可視化軟件專用的格式,必須通過pvm2raw轉換成raw格式,再用vodaconv將之轉換為cube格式才能用VMD顯示。
在這里下載我編譯好的V3程序附帶的pvm2raw程序:/usr/uploads/file/20150611/20150611013430_27584.rar。其中還包含了raw2pvm,用于將raw轉成pvm格式,將在《談談體數據》系列后續文章用到。
將DTI-B0.pvm放到pvm2raw.exe所在的目錄下。建立一個文本文件,內容是pvm2raw DTI-B0.pvm DTI-B0.raw,然后將此文本文件擴展名改為.bat(即Windows下批處理文件)。再雙擊此bat文件執行之,就得到了DTI-B0.raw。
將DTI-B0.raw拷到vodaconv目錄下。由于這個數據庫的體數據都是Big Endian順序,所以應啟動的是vodaconv.exe而非vodaconv_LE.exe,然后依次執行:
DTI-B0.raw
128,128,58
[直接敲回車]
[直接敲回車]
2
2
DTI-B0.cub
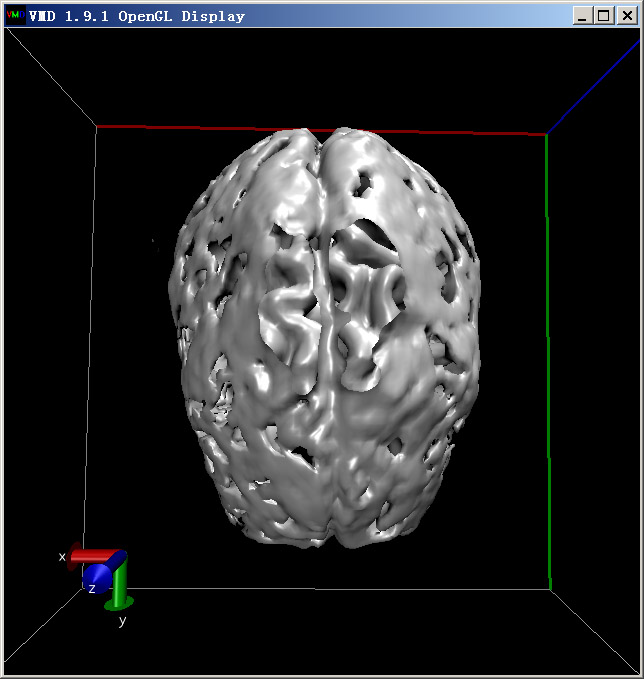
啟動VMD,將DTI-B0.cub拖進VMD main窗口。選Graphics-Representation,將第一個顯示方式的Drawing Method設成Isosurface,Draw從默認的Points改為Solid surface,拖動Isovalue滑條到500左右,就能看到下面的大腦外層輪廓:
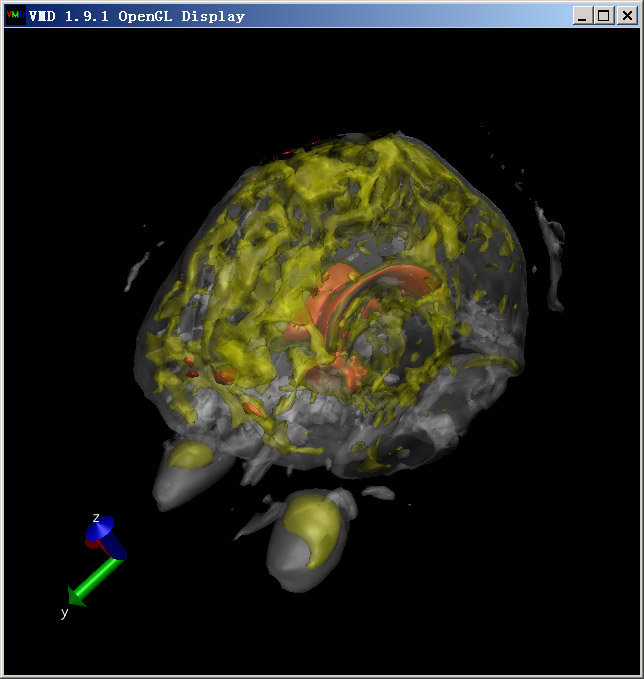
但是這種圖沒法同時看到大腦內外層結構。一種辦法是使用多個不同顏色的對應不同isovalue的透明等值面。先將Display-Rendermode設成GLSL(一些機子上可能因為顯卡太老或驅動問題而選不了,這將導致無法產生透明效果)。在Graphics-Representation里建立三個Isosurface風格的顯示方式,第一個的isovalue設為260,Material設為Transparent;第二個Isosurface也用Transparent,Isovalue設為720,Coloring Method設為ColorID,并選4(黃色);第三個Isosurface用Opaque材質,isovalue設1250,Coloring Method設為ColorID,并選1(紅色)。另外,將這三個顯示方式的Show都從Box+Isosurface改為Isosurface以免盒子邊緣礙眼,將看到如下效果:
也可以用免費的外部渲染器POVray渲染一下,效果會更好:
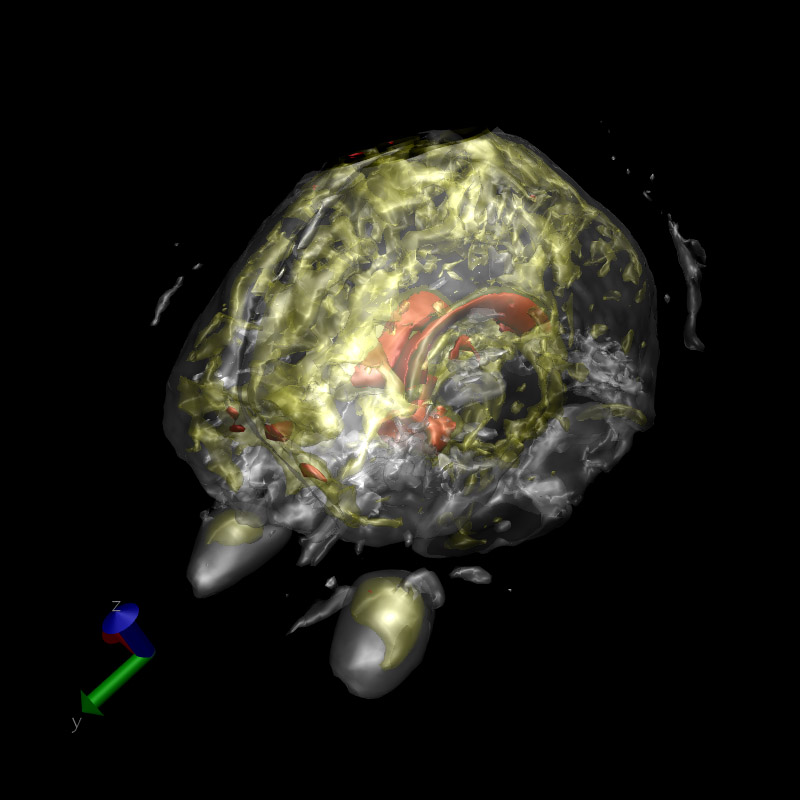
這下,大腦的結構層次就看出來了。最外層等值面會顯示出兩個大橢球,那就是眼珠了。
接下來,我們在VMD里顯示大腦切片圖。實際上,這也就是醫生經常看的大腦切片照片。首先將Display-depth cueing關掉,這樣切片圖會更鮮亮。然后建立一個VolumeSlice的顯示方式,拖動Slice Offset到不同位置,就能看到不同位置的截面圖了。下圖是X的offset值為0.5時的圖
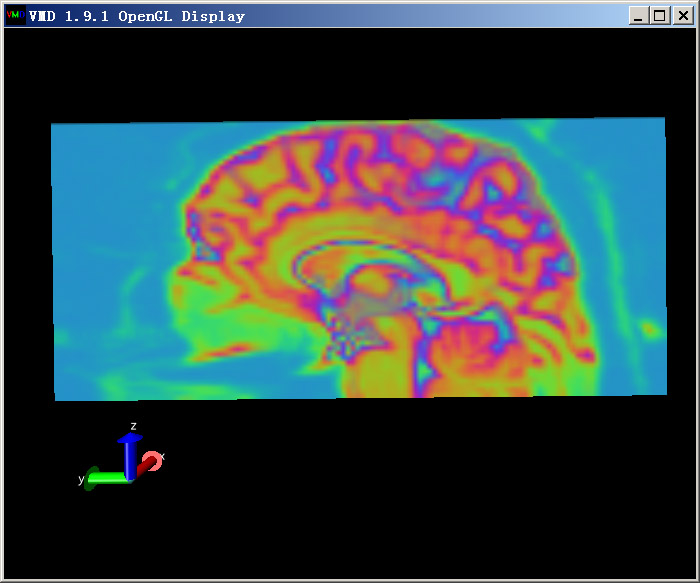
如果想把截面圖弄成黑白的,更有醫院的感覺,可以在Graphics-color-Color scale里將Method設為BlkW,然后將截面圖顯示方式的Coloring Method設為Volume。直接得到的圖像效果不好,對比不明顯,可以在Trajectory標簽頁里調節Color Scale Data Range,比如上下限設為0和1000,然后點Set,效果會好很多。也建議在Display里將所有光源都關掉。得到的Z的offset為0.44的截面圖如下所示:
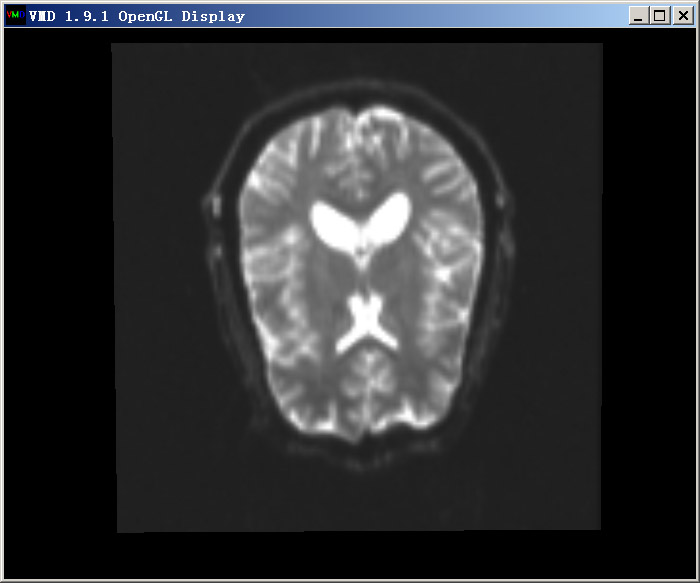
另外,還可以同時建立多個截面顯示方式,同時顯示出大腦不同截面,請自行嘗試。
5 使用Sigmaplot顯示大腦截面圖
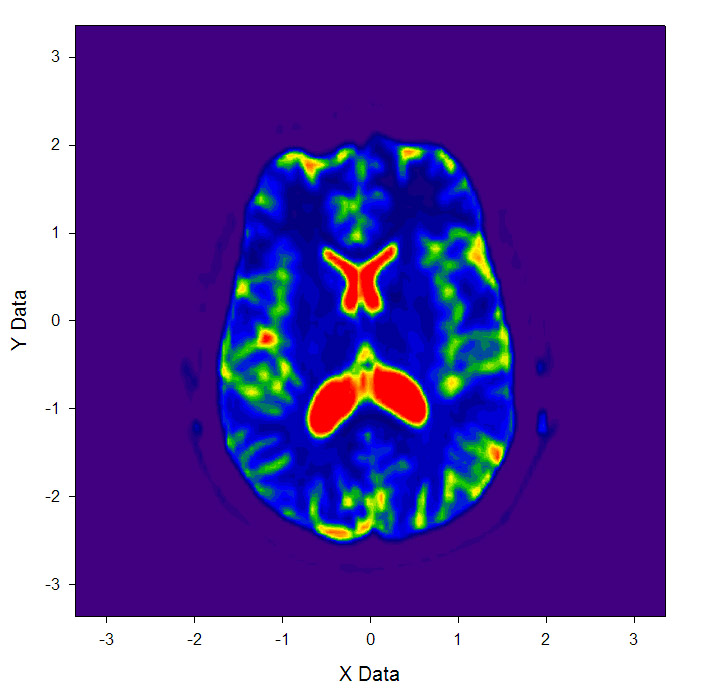
VMD雖然能直接顯示截面圖,但是圖片不夠精細,也不很適合直接拿來作為插圖。一種得到更高質量的截面圖的辦法是用Multiwfn提取出指定平面的數據,然后用Sigmaplot等專業一些的繪圖軟件繪制平面圖。這里,就結合Multiwfn和Sigmaplot繪制上面那張Z的offset為0.44的大腦截面圖。
啟動Multiwfn,輸入DTI-B0.cub的文件路徑,在載入數據結束后會出現格點數據的統計信息。從中可看到這個體數據的z范圍是從-2.85到2.85bohr。VMD中的Z offset=0.44指的是這個XY平面的Z值為整個體數據Z值范圍的44%的位置。因此,我們要提取的是Z值為((2.85*2)*0.44-2.85)*0.5292=-0.181埃的XY平面上的數據。接下來在Multiwfn中輸入
13 //進入主功能13,即格點數據處理功能
2 //提取某個XY平面
-0.181 //Z值(埃)
此時,與Z=-0.181埃最接近的XY的平面上的數據都被輸出到了當前目錄下的output.txt里了。
啟動sigmaplot(本文用的是11.1版),File-import file,選output.txt,然后點import按鈕。導入完畢后,在界面左側找到contour plot按鈕,點擊后再點擊彩色的Filled color按鈕,選XYZ Triplet,X,Y,Z分別設為第1、2、4列,點“完成”。在圖像上雙擊,在Plots-Scale里將上下限分別設為100和1100以改善顯示效果,點“確定”后就會看到下面的圖像,比起VMD顯示的精細很多:
6 總結
通過本文的實例,顯示了計算化學可視化軟件不光可以觀看化學相關的體數據,即分子軌道、電子定域化函數、電子密度等,還可以像通用的體數據可視化軟件一樣研究其它各種類型的體數據,這直接溝通了化學軟件與生活中的物件以及生命醫學間的橋梁。有興趣的話,不妨利用本文的方法自行從那些數據庫中下載感興趣的體系來觀看一下。不過,化學上的軟件在顯示體數據方面比起專業的軟件還顯得比較業余,功能、效率和畫面效果還有不小差距。在《談談體數據》后面的文章中,將介紹利用一些更專業、強大的通用的體數據可視化軟件來繪制出很炫的化學相關的圖形。